En todas las ediciones de mi libro sobre las maravillosas herramientas CRISPR, pero especialmente en la última publicada hasta el momento de Editando genes: recorta, pega y colorea (NextDoor Publishers, marzo de 2021, 3ª edición) me he dedicado a resaltar hacia donde creía que el universo CRISPR bascularía. Cuáles serían las variantes o aplicaciones que podrían triunfar. Y entre ellas siempre he destacado a los editores de bases, una genialidad surgida de la mente inquieta y el talento de David Liu (Instituto BROAD-MIT), uno de los investigadores más imaginativos del panorama CRISPR, responsable no solo de imaginar los editores de bases, sino también la edición PRIME (de calidad), que también ha generado unas enormes perspectivas terapéuticas.
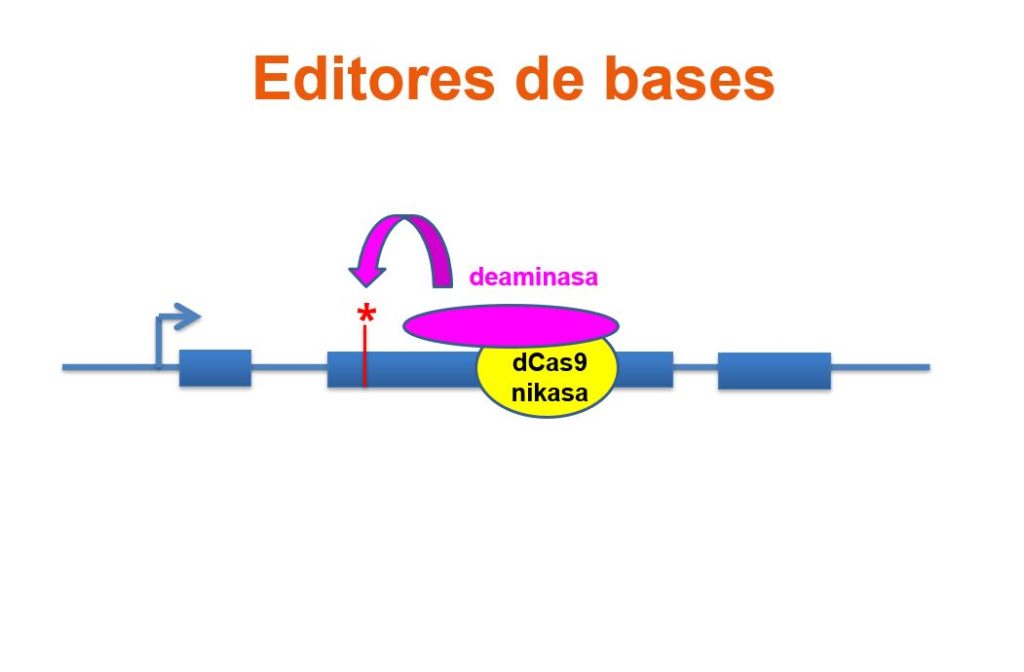
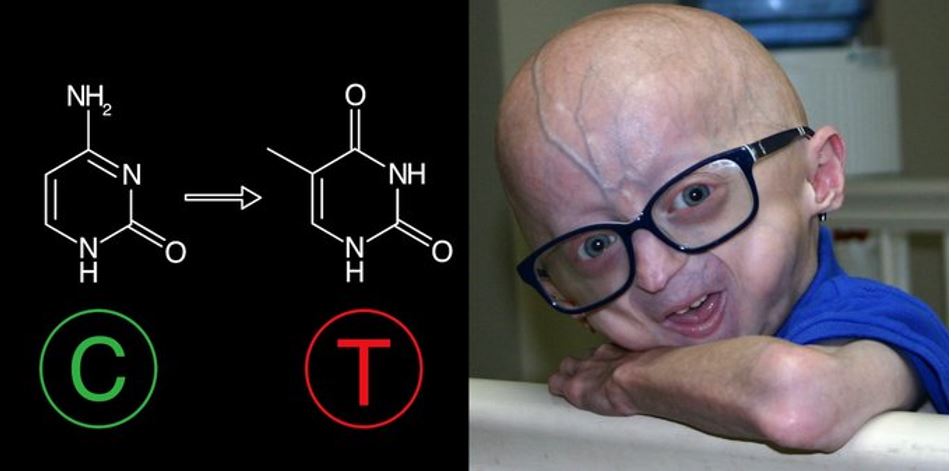
Corría mayo de 2016, menos de cuatro años después de que Emmanuelle Charpentier y Jennifer Doudna hubieran lanzado su propuesta de convertir el sistema de defensa CRISPR que usan procariotas (bacterias y arqueas) para defenderse de virus y plásmidos, cuando David Liu daba la campanada presentando los primeros editores de bases en sociedad. La idea, cuando la leías, era bien simple. Lo difícil era imaginarla antes que nadie te la contara. Se trataba de utilizar variantes de la nucleasa Cas9 total o parcialmente inactivas (dCas9=Cas9 muerta, incapaz de cortar el ADN; o la nikasa, solamente capaz de cortar una de las dos hebras del ADN) para llevar el complejo CRISPR-Cas a nuestro gen favorito (aprovechando la guía de ARN específica que dirigía la Cas9 al sitio complementario en el genoma). La gran diferencia estaba en unir la dCas9 o nikasa a dominios deaminasa, capaces de convertir químicamente la identidad de las bases nitrogenadas (una C en una T, o una A en una G), sin mediar corte de ADN en doble cadena. Esto último era relevante y el mayor talón de Aquiles de la tecnología CRISPR tradicional. Esto es, tras el corte en doble cadena los sistemas de reparación endógenos celulares tienden a restaurar de inmediato la continuidad física del cromosoma, mayoritariamente usando la estrategia denominada NHEJ (unión de extremos no homólogos) que progresa mediante la adición y substracción de nucleótidos al azar, esperando que la suerte lleve a disponer alguna A delante de T o alguna G delante de C para que se establezca el apareamiento descrito por Watson y Crick en 1953 que todos conocemos y es la base del mantenimiento de la información genética en nuestro genoma (y en el de todos los seres vivos, exceptuando algunos bacteriófagos) : la A (adenina) se aparea siempre con la T (timina), y la G (guanina) se aparea siempre con la C (citosina).
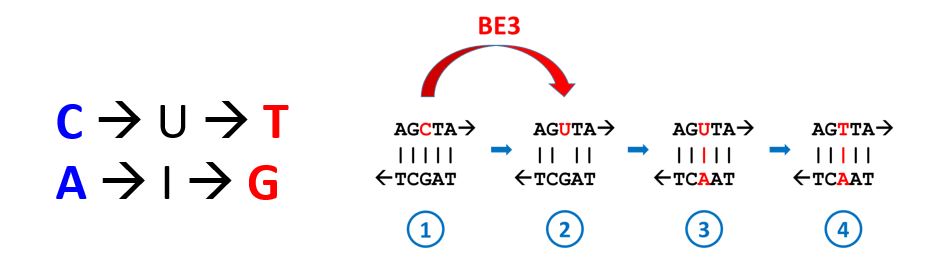
La primera versión de un editor de bases (BE1) fue un CBE (citosine base editor), una dCas9 asociado a una actividad citidina deaminasa, capaz de convertir químicamente una C (citosina) en U (uracilo). Posicionando la guía ARN y la dCas9 de tal manera que el tamaño de la citidina deaminasa fuera a contactar con la C deseada (toda una hazaña a nivel molecular) funcionaba, pero no de forma muy eficiente. Existe una actividad en las células, la Uracilo-ADN-glicosilasa (UDG) que reconocía la U que aparecía desapareada en el ADN y la eliminaba, iniciando la reparación tomando como molde el nucleótido de la otra cadena, una G, y volviendo a poner la C original, con lo cual se volvía a la casilla de salida. Liu inventó una manera de superar este problema añadiendo una tercera actividad al complejo del editor de bases (además de la dCas9 y la citidina deaminasa). Añadió un inhibidor de la Uracilo-ADN-glicosilasa (UGI), y creó la segunda versión: BE2, que mostraba una eficiencia de conversión tres veces mayor que BE1, aunque no superaba un 50%. Para aumentar todavía más el proceso pensó que si era capaz de cortar la cadena complementaria de ADN a la que contenía la base transformada induciría al sistema de reparación a restaurar el corte y entonces tomaría habitualmente la recién transformada U como molde e introduciría una A en la cadena a reparar (la U, como la T, se aparea con la A). Y, finalmente, la A dirigiría en la siguiente ronda de replicación la conversión de la U en T. El resultado fue la tercer versión del editor de bases: BE3. Las tres versiones y sus capacidades descritas en el mismo artículo científico (!).
La propuesta de los editores de bases era rompedora, innovadora, espectacular. Sin embargo no estaba exenta de problemas. La adición de la nikasa, que cortaba una de las cadenas de ADN, reducía pero no hacía desaparecer la posibilidad de generar mosaicismo genético (reducido al 1%), pero seguía mostrando un moderado, pero preocupante, número de potenciales transformaciones en Cs no deseadas (efectos off-target). Al año siguiente David Liu mejoró estos editores de bases usando unas Cas9-nikasa modificadas capaces de aceptar diferentes secuencias PAM (motivo adyacente al protoespaciador) lo cual permitía programar y diseñar una mayor diversidad de guías ARN y llegar a un mayor número de posiciones C en el genoma para ser transformadas en T. Seguidamente aumentó la especifidad de las Cas utilizadas substituyéndolas por sus variantes HF (high fidelity).
A finales de 2017 David Liu daba otra nueva campanada al describir un nuevo tipo de editores de bases, ABE (adenin base editor), substituyendo la actividad citidina deaminasa por otra, creada en el laboratorio, de adenina deaminasa, todo un hallazgo. Si los CBE eran capaces de convertir C en T, los ABE eran ahora capaces de convertir A en G, en este caso a través de inosina (I), por un mecanismo similar. En este primer trabajo se demostró que los ABE eran unos editores de base muy eficientes en convertir A en G, sin apenas problemas de off-targets. Sin embargo, a principios de 2019 aparecieron sendos artículos en la revista Science que daban cuenta de un número excesivo de mutaciones no deseadas en otras partes del genoma (off-targets) cuando se usaban los editores de bases del tipo CBE (pero no del tipo ABE) tanto en animales (ratones) como en plantas (arroz), que enfriaron momentáneamente las expectativas de los editores de bases.
A partir de ahí, el grupo de David Liu desarrolló numerosas mejoras y nuevas variantes cada vez más eficientes y específicas de CBE y ABE, presentando en 2018 las variantes BE4max y ABEmax que empezaron en 2020 a usarse in vivo, en modelos de ratón de enfermedades humanas, como la tirosinemia hereditaria de tipo I o un modelo en ratón de sordera congénita o un modelo de degeneración de retina. Es importante señalar que todas estas múltiples variantes de ABEs y CBEs están disponibles, para su uso académico, en el repositorio de reactivos CRISPR en Addgene.

En mayo de 2018, convencido del gran potencial terapéutico que tendrían los editores de bases, David Liu decide fundar, junto a sus colegas Feng Zhang y J. Keith Joung, otros dos pioneros y figuras igualmente relevantes del universo CRISPR, la empresa BEAM Therapeutics. Y esta empresa será la que marcará a partir de ese momento el ritmo de desarrollo de los editores de bases y sus aplicaciones terapéuticas.
A principios de este año 2021, David Liu volvió a sorprender al mundo comunicando su éxito al usar editores de bases (ABE) para corregir una mutación puntual en el gen lamin A en un modelo animal, en ratones, de la Progeria de Hutchinson-Gilford, una enfermedad extremadamente rara que progresa con envejecimiento acelerado. En este trabajo Liu y colaboradores demostraron que era posible corregir la mutación en células humanas de pacientes en cultivo, y también en ratones portadores de la misma mutación. En ratones mutantes de dos semanas de edad, se les proporcionaba el editor de bases con la guía ARN correspondiente a través de vectores virales adeno-asociados (AAV), con una única inyección retro-orbital y ello era suficiente para conseguir corregir del 20 al 60% de las células en diferentes órganos, suficiente para evitar la aparición de los síntomas característicos de esta patología y para aumentar la vida de estos ratones desde unos 200 días a más de 500 días. Este artículo acercaba los editores de bases al futuro tratamiento de enfermedades en pacientes.
Esta semana pasada acabamos de conocer el penúltimo de los éxitos derivados del uso de editores de bases con finalidad terapéutica. En un artículo publicado en la revista Nature un conjunto de investigadores (Musunuru et al. Nature 2021) suben la apuesta y evalúan el papel de los editores de bases en un modelo animal mucho más cercano a nosotros, en primates no humanos, en macacos. Es la primera vez que se reporta el uso exitoso de editores de bases en monos. Entre los autores hay diversos investigadores de la empresa BEAM Therapeutics, que son quienes han preparado el editor de bases más evolucionado y optimizado que conocemos, hasta el momento, denominado ABE8.8, capaz de substituir una A por una G en lugares precisos del genoma, guiado por ARN, con eficacia y gran fiabilidad. Este editor fue presentado ante la comunidad científica en el mes de julio del año pasado, 2020.

La aproximación terapéutica que realiza este estudio reciente es muy ingeniosa. El gen PCSK9 codifica un inhibidor de la expresión del receptor de las lipoproteínas de baja densidad (LDL), que son las que transportan colesterol por la sangre, y las que asociamos popularmente como marcadoras del «colesterol malo» (mientras que las lipoproteínas de alta densidad, HDL, serían las que se asocian a marcadoras del «colesterol bueno«). Normalmente, la actividad de PCSK9 reduce la cantidad del receptor de LDL por parte de los hepatocitos del hígado y tiene como consecuencia un aumento de las LDL circulantes por la sangre, un aumento del colesterol, lo cual acarrea riesgos elevados de patologías cardiovasculares como la formación de placas de ateroma (aterosclerosis), que es una de las enfermedades causantes de un mayor número de muertes en todo el mundo. La inactivación del gen PCSK9 elimina el inhibidor y entonces aumenta la expresión del receptor de LDL, que es capaz de captar mayor número de LDL circulantes, disminuyendo la cantidad de estas, y, por lo tanto, del colesterol el sangre. Aproximadamente entre un 2-3% de personas son mutantes de forma natural en este gen, y tienen por lo tanto riesgo muy bajo de desarrollar enfermedades cardiovasculares como la aterosclerosis, sin problemas de salud adicionales.
Teniendo en cuenta todo lo anterior Musunuru y colaboradores desarrollaron un editor de bases encaminado a introducir una mutación (A–>G) en la señal de procesamiento del primer intrón del gen PCSK9 (primero en ratones y luego en macacos), con objeto de generar un ARN mensajero incorrectamente procesado que sustentaría la traducción de una proteína PCSK9 errónea. Y efectivamente es lo que consiguieron. Una sola administración sistémica (vía intravenosa) de este editor de bases y su guía correspondiente en formato ARN, protegido por nanopartículas lipídicas, y aprovechando el conocimiento de las modificaciones en los ribonucleósidos para estabilizar las moléculas de ARN que se ha aplicado a las vacunas ARNm contra la COVID-19, sirvió para editar e inactivar el gen PCSK9 en macacos en un 90% de las células, mayoritariamente del hígado, y con ello reducir la cantidad de LDL circulantes en un 60%, valores que se mantuvieron 8-10 meses después del tratamiento, con unos valores muy reducidos de off-targets en los animales. Es un resultado espectacular, realizado ya en un animal muy parecido a nosotros, que abre las puertas a próximos ensayos clínicos que puedan llevar los beneficios terapéuticos de los editores de bases para tratar enfermedades metabólicas complejas en seres humanos.
Un equipo competidor obtuvo resultados similares, también en primates no humanos, aunque menos espectaculares, probablemente porque usaron un editor de bases (ABEmax), desarrollado en 2018, y por ello anterior al usado por Musunuru, quien contó con la colaboración de los investigadores de la empresa BEAM Therapeutics que le permitió usar el mejor y más moderno editor de bases conocido: ABE8.8.
¡Atentos a los editores de bases! Llegaron unos años después que las herramientas CRISPR-Cas9 tradicionales de edición genética, pero están llamados a superarlas, en eficacia, precisión y seguridad. Y, por ello, no es descartable que veamos llegar pronto estas innovadoras variantes del sistema CRISPR, imaginadas por David Liu, a los ensayos clínicos y, quién sabe, quizás en un futuro esperemos que próximo para el tratamiento de muchas enfermedades humanas, hoy en día incurables.
Finalmente una reflexión, recuerda que los sistemas CRISPR-Cas9, por sí mismos, no son herramientas terapéuticas, solamente cortan el ADN y propician su reparación o edición, que es donde aparece la terapia génica. Por el contrario los editores de bases sí son herramientas terapéuticas. Ellos mismos son capaces de substituir con precisión y eficacia los nucleótidos erróneos o los que sea necesario mutar, directamente. Una gran diferencia.


Gran artículo Lluís! Muy bien explicado para que todos seamos capaces de entender el potencial de los editores de bases.
Somos muchas las personas que tenemos puestas muchas esperanzas en los editores de bases. Creo que uno de los grandes retos a superar es el delivery a órganos como el cerebro. ¿Cómo lo ves tú?¿Hay algún caso de éxito de edición en el cerebro?
Muchas gracias Ana. Hay muchos retos todavía por solventar. El que mencionas (entrega en el sistema nervioso central) es uno de ellos. Existen vectores adenoasociados capaces de cruzar la barrera hematoencefálica, eso podría ser una solución, ya explorada con herramientas CRISPR clásicas en modelos animales.
Buenas tardes amigo Montoliu, soy un fiel seguidor suyo Ud. a cambiado mi forma de pensar sobre la genética solo soy un simple payasito que le gusta estar al dia en los avances cientificos un saludo desde Pando, Bolivia desde lo mas profundo de la selva
Cordiales saludos y gracias!
Saludos, muy interesante y muy bien explicado. Sabe si el gen de iPCSK9 está cerca de oncogenes? ¿O hay alguna posibilidad de activarlos ‘sin querer’ usando esta tecnología?
Fenomenal artículo e información Sr.Montoliu.
Ojalá pronto podamos ver el uso de estos editores en la corrección de enfermedades genéticas hereditarias.
En mi caso se trata de una Neuropatia óptica similar a la Neuropatia óptica de Leber, que produce la atrofia bilateral del nervio óptico.
La mutación que la produce está en el gen mitocondrial MT-CYB, donde hay el cambio nucleotídico m.15674T>C, produciendo un cambio de proteina Ser310Pro.
Gracias por potenciar la investigación acerca de estos temas genéticos, es primordial!!!!
Un saludo cordial
Ojalá los avances sean rápidos en este campo para que las terapias puedan llegar pronto a los pacientes.