Cuando en febrero de 2001 se publicó finalmente el primer borrador del genoma humano uno de los datos que más desconcertaron y descolocaron a los científicos fue el exíguo número de genes que parecíamos tener. Las primeras estimaciones hablaban de 30.000-40.000 genes codificantes (esto es, que portan información genética codificada que se puede transformar en proteínas). Las apuestas entre los genetistas suponían que nuestro genoma tendría unos 100.000 genes. Pero cuál fue nuestra sorpresa cuando comprobamos, a medida que se fue corrigiendo y puliendo la secuencia de ADN del genoma humano, que teníamos apenas una quinta parte de ese número de genes, un valor muy similar al número de genes que posee el genoma de un gusano, del nemátodo (Caenorhabitis elegans) que mide ~1 mm y que tiene alrededor de 1.000 células (exactamente 959), frente a los aproximadamente 30 billones de células que tiene un ser humano. ¿Pero cómo podemos tener los mismos genes que un gusano que apenas vemos a simple vista? Vayamos por partes.
Tras definir el tamaño del genoma humano esta nueva entrada del blog la dedicaré a delimitar el número de genes que tenemos en el genoma humano. Naturalmente para cuantificar algo lo primero que tenemos que hacer es definir lo que queremos contabilizar. En otras palabras: ¿qué entendemos por gen? Pues bien, aquí ya empezamos con las variaciones que coexisten en biología. Históricamente los genes se definían como «fragmentos de ADN que portan información genética codificada para producir una proteína«. Pero pronto descubrimos que los genes no eran contiguos, que existían intrones que separaban los exones, y que tenían elementos reguladores, cercanos y/o muy alejados, que eran imprescindibles para su correcta expresión. Entonces, una definición de gen actualizada podría ser: «un intervalo de ADN en el cromosoma que se transcribe en forma de ARN y luego se traduce en forma de una proteína funcional«. Pero esta definición deja fuera a muchos genes que sabemos que no completan este camino, y solamente se transcriben a moléculas ARN, que son funcionales por sí solas y actúan de diferentes maneras, activando o reprimiendo otros genes (como los ARNs de pequeño [miRNA] y de gran tamaño [lncRNA] no codificantes) o los que son esenciales para la síntesis de proteínas (como los ARN ribosomales [rRNA] o los ARN de transferencia [tRNA]). Por ello, una definición universal más inclusiva sería hablar de «un intervalo de ADN que se transcribe y da lugar a moléculas de ARN funcional«, lo cual incluiría los genes que codifican proteínas y los que no.
A los efectos de la cuantificación de genes en genomas el acuerdo actual, de nuevo arbitrario, es contar separadamente los genes codificantes (los que portan información que acaba traduciéndose en forma de proteínas) y los genes no codificantes (los que se transcriben a ARN y actúan como ARN, sin traducirse a proteínas). Adicionalmente, cuando nos referimos al número de genes de un organismo solemos referirnos a la primera de estas categorías, solamente teniendo en cuenta los genes codificantes.
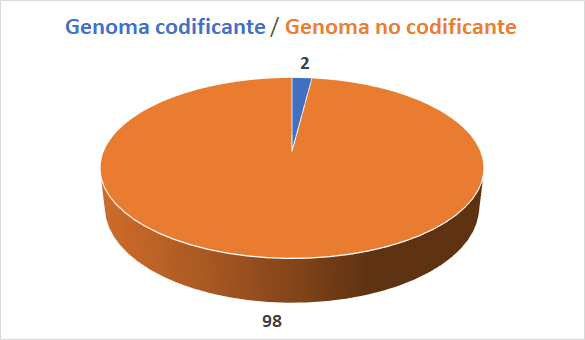
Si juntamos todos los segmentos de ADN que corresponden a genes codificantes, que se transcriben a ARN y se traducen a proteína, el resultado será, aproximadamente un 2% del tamaño total de nuestro genoma (de referencia). O sea, de los 3.272.116.950 pares de bases que tiene el genoma de referencia solamente un 2% correspondería a genoma codificante, es decir: 65.442.339 pares de bases. ¿Y que hay en el 98% restante que corresponde al genoma no-codificante? Pues muchos elementos reguladores, que determinan cuando y dónde se deberá expresar un gen, también están aquí todos los genes no codificantes (miRNA, lncRNA, rRNA y tRNA), diferentes familias de elementos móviles, transposones y retrotransposones, y múltiples familias también de ADN repetitivo (satélite). También encontramos los pseudogenes, generalmente correspondientes a secuencias parciales o totales de genes que han quedado inactivos a lo largo de la evolución al acumular numerosas mutaciones. Hay diferentes tipos de pseudogenes. Suelen originarse a partir de genes, de otros fragmentos de ADN o mediante retrotranscripción de ARN mensajeros, mediante la accción de una transcriptasa inversa, y acaban insertados en diferentes partes del genoma. Generalmente los pseudogenes no son funcionales. Por el contrario, el proyecto internacional ENCODE determinó que más del 80% de nuestro genoma sería funcional desde el punto de vista bioquímico, lo cual supera ampliamente el 2% que solamente contempla la parte codificante y concede al genoma no codificante una relevancia insoslayable.
Pero volvamos al genoma codificante, y determinemos el número de genes que tiene nuestro genoma. Por supuesto me refiero al genoma de referencia, el que contiene una copia de cada uno de los 22 cromosomas autosómicos, un cromosoma X, un cromosoma Y, el genoma mitocondrial y las secuencias de ADN no ensambladas. Primero mediante procedimientos bioinformáticos y luego, en la mayor parte de los genes, mediante evidencias experimentales, se ha logrado encontrar cuántos genes tenemos en cada cromosoma. Veamos la siguiente tabla.
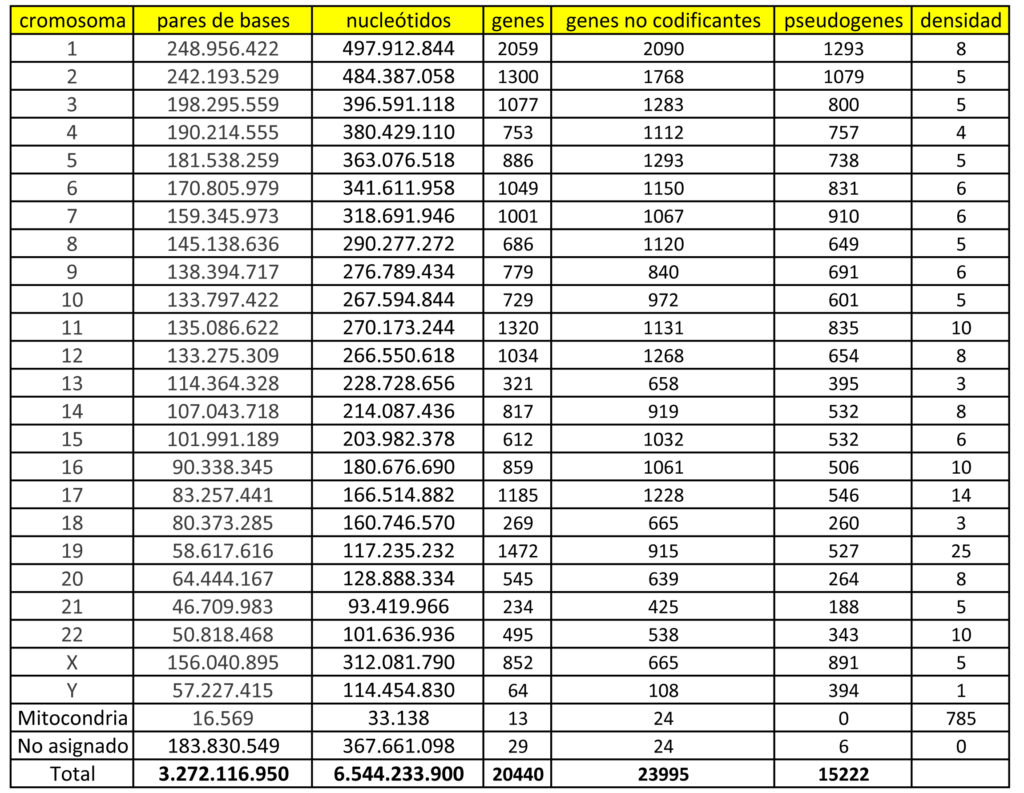
Como se puede apreciar en la tabla adjunta, generalmente cada uno de nuestros cromosomas tiene unos 2.000 genes o menos. El que más tiene es el cromosoma 1 (2.059 genes), que además es el cromosoma de mayor tamaño. Y el que menos tiene es el cromosoma Y (64 genes), uno de los de menor tamaño, teniendo en cuenta que el ADN mitocondrial contiene 13 genes y en las secuencias no ensambladas encontramos unos 29 genes. No todos los cromosomas son igual de «densos» en número de genes. El cromosoma Y y el cromosoma 13 son los que contienen, en proporción a su tamaño, una menor densidad de genes, mientras que el cromosoma que contiene más genes por unidad de tamaño (por 1.000.000 de pares de bases) es el cromosoma 19. Si consultáis la tabla veréis que el cromosoma 19 tiene 25 veces más número de genes por millón de pares de bases que el cromosoma Y.
En total nuestro genoma de referencia contiene, actualmente, 20.440 genes. Un número muy alejado de las primeras estimaciones de 100.000 genes que se esperaban. Por eso, habitualmente la cifra aproximada que se usa para referirse al número de genes del genoma humano es de ~20.000 genes. Calculemos ahora el número de genes que tiene una persona de sexo masculino (46XY) o los genes que tiene una persona de sexo femenino (46XX).
Para calcular el número de genes de una persona de sexo masculino (46XY) sumaremos los genes de cada uno de los 22 cromosomas autosómicos, los que tiene un cromosoma X, los que tiene un cromosoma Y, los mitoconcriales y los no ensamblados, y, en este caso, nos saldrá el mismo número de genes que el genoma de referencia: 20.440 genes.
Para calcular el número de genes de una persona de sexo femenino (46XX) sumaremos los genes de cada uno de los 22 cromosomas autosómicos, los que tiene un cromosoma X, los mitoconcriales y los no ensamblados, y, en este caso, nos saldrá, lógicamente (al no contabilizar el cromosoma Y) un número de genes inferior, exactamente: 20.376 genes.
Por lo tanto, los individuos 46XX a pesar de tener un genoma de mayor tamaño que los individuos 46XY tienen un número algo inferior de genes, exactamente 64 genes menos, los que corresponden al cromosoma Y. Es decir, a pesar que las personas de sexo femenino pueden disponer de un genoma más grande este es por contra menos diverso genéticamente, al faltarles los 64 genes adicionales que solamente encontramos en las personas de sexo masculino.
Adicionalmente, nuestro genoma de referencia contiene 23.995 genes no codificantes y 15.222 pseudogenes.
Los genes del genoma humano en relación a otras especies
Ahora veamos: ¿esto de tener unos 20.000 genes es mucho o es poco? ¿Hay especies de animales o plantas que tienen más o menos genes que nosotros? ¿Somos la especie que más genes tiene? Vamos a revisar la siguiente tabla, en la que he incorporado el número de genes conocidos y el tamaño del genoma de referencia (en pares de bases, a partir de los valores disponibles, dado que en algunos casos el genoma no está completo y, por ejemplo, las regiones repetitivas no están totalmente incorporadas) de cien (100) especies de animales, plantas, hongos y bacterias, a partir de los datos de ENSEMBL (datos obtenidos de la web de ENSEMBL el 29 de noviembre de 2020). La densidad vuelve a ser el mismo parámetro usado en la tabla anterior y da idea de la cantidad de genes de la especie en relación a su genoma (número de genes / millón de pares de bases). Valores altos indican genomas muy compactos. Valores más bajos sugieren genomas con mayor proporción de regiones no codificantes, más espaciados.
La tabla está ordenada atendiendo al número de genes de cada especie, de mayor a menor.
| Posición | organismo | especie | genes | genoma | densidad |
| 1 | trigo | Triticum aestivum | 107.891 | 14.547.261.565 | 7 |
| 2 | colza | Brassica napus | 101.040 | 738.357.821 | 137 |
| 3 | soja | Glycine max | 55.897 | 955.377.461 | 59 |
| 4 | carpa dorada | Carassius auratus | 54.774 | 1.820.635.050 | 30 |
| 5 | girasol | Helianthus annuus | 52.191 | 2.925.295.703 | 18 |
| 6 | olivo | Olea europaea var. sylvestris | 50.681 | 1.140.989.389 | 44 |
| 7 | salmón | Salmo salar | 47.329 | 3.412.130.248 | 14 |
| 8 | cerezo | Prunus avium | 43.349 | 272.361.615 | 159 |
| 9 | trucha | Oncorhynchus mykiss | 41.365 | 2.178.999.613 | 19 |
| 10 | álamo negro | Populus trichocarpa | 41.335 | 422.940.594 | 98 |
| 11 | arroz | Oryza sativa Indica | 40.745 | 411.710.190 | 99 |
| 12 | manzano | Malus domestica | 40.624 | 702.961.352 | 58 |
| 13 | trébol | Trifolium pratense | 39.948 | 296.217.290 | 135 |
| 14 | cebada | Hordeum vulgare | 39.841 | 8.059.674.078 | 5 |
| 15 | maíz | Zea mays | 39.591 | 2.104.350.183 | 19 |
| 16 | patata | Solanum tuberosum | 39.021 | 727.424.546 | 54 |
| 17 | plátano | Musa acuminata | 36.525 | 390.578.572 | 94 |
| 18 | guindilla | Capsicum annuum | 35.845 | 3.063.864.880 | 12 |
| 19 | tomate | Solanum lycopersicum | 34.658 | 827.747.456 | 42 |
| 20 | zanahoria | Daucus carota | 32.109 | 421.502.825 | 76 |
| 21 | pistacho | Pistacia vera | 31.784 | 671.152.441 | 47 |
| 22 | vid | Vitis vinifera | 29.971 | 486.175.922 | 62 |
| 23 | melón | Cucumis melo | 28.299 | 357.857.370 | 79 |
| 24 | judía | Phaseolus vulgaris | 28.134 | 472.453.824 | 60 |
| 25 | almendro | Prunus dulcis | 27.966 | 227.498.357 | 123 |
| 26 | arabidopsis | Arabidopsis thaliana | 27.655 | 135.670.229 | 204 |
| 27 | marihuana | Cannabis sativa | 27.249 | 736.163.757 | 37 |
| 28 | melocotón | Prunus persica | 26.873 | 224.638.928 | 120 |
| 29 | piña | Ananas comosus | 26.730 | 315.839.004 | 85 |
| 30 | remolacha | Beta vulgaris | 26.521 | 517.420.803 | 51 |
| 31 | cardo | Cynara cardunculus | 26.505 | 724.667.265 | 37 |
| 32 | pez cebra | Danio rerio | 25.592 | 1.674.207.132 | 15 |
| 33 | café | Coffea canephora | 25.574 | 471.313.922 | 54 |
| 34 | dorada | Sparus aurata | 25.222 | 833.578.411 | 30 |
| 35 | mandarina | Citrus clementina | 25.000 | 295.129.112 | 85 |
| 36 | pepino | Cucumis sativus | 23.780 | 193.829.320 | 123 |
| 37 | pez payaso | Amphiprion ocellaris | 23.592 | 880.720.895 | 27 |
| 38 | chimpancé | Pan troglodytes | 23.534 | 3.385.800.935 | 7 |
| 39 | armadillo | Dasypus novemcinctus | 22.711 | 3.299.882.059 | 7 |
| 40 | sandía | Citrullus lanatus | 22.541 | 365.450.462 | 62 |
| 41 | ratón | Mus musculus | 22.519 | 3.486.944.526 | 6 |
| 42 | rata | Rattus norvegicus | 22.250 | 3.042.335.753 | 7 |
| 43 | vaca | Bos taurus | 21.880 | 2.715.853.792 | 8 |
| 44 | gorila | Gorilla gorilla gorilla | 21.794 | 2.917.385.452 | 7 |
| 45 | macaco | Macaca mulatta | 21.761 | 2.971.331.530 | 7 |
| 46 | fugu | Takifugu rubripes | 21.411 | 384.110.215 | 56 |
| 47 | cabra | Capra hircus | 21.361 | 2.922.813.246 | 7 |
| 48 | cerdo | Sus scrofa | 21.303 | 2.478.444.698 | 9 |
| 49 | rodaballo | Scophthalmus maximus | 21.000 | 524.979.463 | 40 |
| 50 | caballo | Equus caballus | 20.955 | 2.506.966.135 | 8 |
| 51 | hámster chino | Cricetulus griseus | 20.824 | 2.323.924.942 | 9 |
| 52 | león marino | Zalophus californianus | 20.818 | 2.408.641.788 | 9 |
| 53 | conejo | Oryctolagus cuniculus | 20.612 | 2.604.023.284 | 8 |
| 54 | oveja | Ovis aries | 20.506 | 2.869.914.396 | 7 |
| 55 | hombre | Homo sapiens | 20.440 | 3.272.116.950 | 6 |
| 56 | orangután | Pongo abelii | 20.424 | 3.109.347.532 | 7 |
| 57 | perro | Canis lupus familiaris | 20.257 | 2.392.715.236 | 8 |
| 58 | gusano | Caenorhabditis elegans | 20.191 | 103.022.290 | 196 |
| 59 | bacalao | Gadus morhua | 20.095 | 608.029.870 | 33 |
| 60 | elefante | Loxodonta africana | 20.033 | 3.118.565.340 | 6 |
| 61 | rana africana | Xenopus tropicalis | 19.987 | 1.440.398.454 | 14 |
| 62 | asno | Equus asinus asinus | 19.963 | 2.321.044.345 | 9 |
| 63 | hurón | Mustela putorius furo | 19.910 | 2.277.906.570 | 9 |
| 64 | koala | Phascolarctos cinereus | 19.833 | 3.192.581.492 | 6 |
| 65 | cachalote | Physeter catodon | 19.717 | 2.512.132.974 | 8 |
| 66 | gato | Felis catus | 19.587 | 2.491.009.630 | 8 |
| 67 | león | Panthera leo | 19.550 | 2.406.807.619 | 8 |
| 68 | ballena azul | Balaenoptera musculus | 19.518 | 2.379.995.981 | 8 |
| 69 | dromedario | Camelus dromedarius | 19.406 | 2.154.386.959 | 9 |
| 70 | panda | Ailuropoda melanoleuca | 19.343 | 2.245.312.831 | 9 |
| 71 | oso polar | Ursus maritimus | 18.724 | 2.301.379.344 | 8 |
| 72 | ardilla | Ictidomys tridecemlineatus | 18.474 | 2.311.076.758 | 8 |
| 73 | conejo de indias | Cavia porcellus | 18.095 | 2.663.369.733 | 7 |
| 74 | cobra | Naja naja | 18.011 | 1.768.535.092 | 10 |
| 75 | tigre | Panthera tigris altaica | 17.544 | 2.332.832.693 | 8 |
| 76 | ornitorrinco | Ornithorhynchus anatinus | 17.418 | 1.858.552.590 | 9 |
| 77 | gallo | Gallus gallus | 16.878 | 1.136.292.815 | 15 |
| 78 | pato | Anas platyrhynchos platyrhynchos | 16.618 | 1.136.415.614 | 15 |
| 79 | delfín | Tursiops truncatus | 16.550 | 2.298.460.478 | 7 |
| 80 | ganso | Anser cygnoides | 15.439 | 1.130.276.682 | 14 |
| 81 | canario | Serinus canaria | 15.225 | 1.152.083.301 | 13 |
| 82 | pavo | Meleagris gallopavo | 14.123 | 935.922.388 | 15 |
| 83 | mosca del vinagre | Drosophila melanogaster | 13.947 | 142.573.024 | 98 |
| 84 | mochuelo | Athene cunicularia | 13.835 | 1.157.069.330 | 12 |
| 85 | musaraña | Sorex araneus | 13.187 | 1.832.864.697 | 7 |
| 86 | lamprea | Petromyzon marinus | 10.415 | 647.368.134 | 16 |
| 87 | levadura | Saccharomyces cerevisiae | 6.600 | 12.157.105 | 543 |
| 88 | Pseudomonas putida | Pseudomonas putida | 5.861 | 6.484.062 | 904 |
| 89 | Bacillus thuringiensis | Bacillus thuringiensis | 5.698 | 5.799.126 | 983 |
| 90 | Bacillus anthracis | Bacillus anthracis | 5.317 | 5.490.124 | 968 |
| 91 | Yersinia pestis | Yersinia pestis | 4.268 | 4.714.480 | 905 |
| 92 | Escherichia coli | Escherichia coli K12 | 4.051 | 4.558.660 | 889 |
| 93 | Mycobacterium tuberculosis | Mycobacterium tuberculosis | 4.040 | 4.327.834 | 933 |
| 94 | Bacillus subtilis | Bacillus subtilis | 3.940 | 4.263.792 | 924 |
| 95 | Vibrio cholerae | Vibrio cholerae | 3.585 | 4.012.216 | 894 |
| 96 | Listeria monocytogenes | Listeria monocytogenes | 3.131 | 2.776.517 | 1.128 |
| 97 | Legionella pneumophila | Legionella pneumophila | 3.105 | 3.470.989 | 895 |
| 98 | Staphylococcus aureus | Staphylococcus aureus | 2.500 | 2.787.689 | 897 |
| 99 | Streptococcus thermophilus | Streptococcus thermophilus | 2.037 | 1.861.792 | 1.094 |
| 100 | Streptococcus pyogenes | Streptococcus pyogenes A20 | 1.828 | 1.837.281 | 995 |
De esta tabla se puede destacar lo siguiente, entre otras muchas conclusiones que pueden derivarse:
- La especie humana es más bien normalita. De estas 100 especies de animales, plantas y bacterias ocupa la zona media, la posición 55. Hay muchas especies animales (p.e. el ratón) que tienen muchos más genes que nosotros y bastantes también que tienen menos, pero para nada somos singulares en este aspecto. Tenemos prácticamente los mismos genes que el gusano C. elegans, a pesar de ser muy diferentes en todo: tamaño, ciclo de vida, número de células… Una buena cura de humildad si esperábais que nuestra especie destacara en tamaño de genoma o en número de genes. No es el caso. Tenemos aproximadamente el mismo número de genes que una oveja.
- Las bacterias son los organismos que tienen menos genes y también son los que tienen los genomas más pequeños y compactos. Las bacterias de la tabla oscilan entre apenas 1.828 genes, que son los que necesita Streptococcus pyogenes (causante de otitis y laringitis) para vivir intracelularmente y los casi 6.000 que tiene Pseudomonas putida, una bacteria típica del medio ambiente. Con los genomas tan reducidos también son las bacterias los organismos que tienen mayor densidad génica, alrededor de 1.000 genes por millón de pares de bases.
- La levadura del pan, cerveza y vino (Saccharomyces cerevisiae), que es un organismo eucariota, un hongo, relativamente simple, apenas tiene 739 genes más que un organismo procariota como Pseudomonas putida.
- Las aves tienen habitualmente menos de 17.000 genes, menos que muchos otros animales y un número bastante parecido al de los reptiles (cobra), con quienes están evolutivamente emparentadas.
- La mayor parte de animales terrestres y mamíferos acuáticos presentan alrededor de 20.000 genes (como los humanos). El mayor número de genes de la tabla le corresponde al chimpancé, con 23.534 genes, y el menos número corresponde a la musaraña, con 13.187 genes.
- Los peces suelen presentar un número mayor de genes, de 20.095 genes que tiene el bacalao a los 54.774 que tiene la carpa dorada (los peces rojos de todos los estanques y de los acuarios).
- Las plantas son los organismos que contienen habitualmente un número mayor de genes, desde los 22.541 genes de la sandía hasta los 107.891 del trigo. Sin embargo en las plantas se dan fenómenos de hibridación y duplicación de genomas que explican, en parte, estos números tan elevados. El trigo es hexaploide, producto de la hibridación de tres genomas, con alrededor de 33.000 genes aportados por cada una de las tres especies ancestrales. La colza, con sus 101.040 genes es producto de la hibridación de dos especies ancestrales seguido de una duplicación de todos los cromosomas, un proceso conocido por alopoliploidía, lo que sugeriría que cada una de las dos especies ancestrales aportó alrededor de 25.000 genes.
- El genoma más compacto de todas las plantas es el de la especie modelo Arabidopsis thaliana, con una densidad de 204 genes por millón de pares de bases.
- El genoma más compacto de todos los animales es el del pez fugu, con una densidad de 56 genes por millón de pares de bases. Esta especie fue propuesta por ello como especie modelo al contar con la menor proporción de genoma no codificante, además de ser una verdadera delicatessen en Japón, cuyo consumo seguro exige cocineros especializados que sepan prepararlo eliminando el hígado y glándulas anexas que son extremadamente tóxicas, mortales, para quienes lo consumen.
- El animal con un menor número de genes es la lamprea, con apenas 10.415 genes. La lamprea no es un pez, es un fósil viviente, de la superclase ciclostomata. Estos animales se sitúan en el origen de los vertebrados. Se alimentan parasitando otros animales, otros peces y mamíferos marinos, habitualmente.
- La planta con un menor número de genes es la sandía, que tiene 22.541 genes y que además tiene un genoma razonablemente compacto también.
- Los seres vivos se han diversificado, evolucionado y aprendido a sobrevivir con una amplia variación en el número de genes, que oscilan entre los 1.828 de la bacteria patógena Streptococcus pyogenes, y los 55.897 de la soja, lo cual representa una variación de más de 30 veces. Esta bacteria, y otras todavía más austeras en el número de genes, nos indican el mínimo conjunto de genes que necesita una célula procariota/eucariota para sobrevivir. La bacteria Carsonella rudii simbiótica con insectos que se alimentan de la savia de las plantas es la que necesita menos genes para sobrevivir, exactamente 182 genes solamente.
¿El número de genes que tiene una especie nos da idea de la complejidad de la misma?
La verdad es que no. Aunque podría parecer que cuantos más genes tiene una especie más funciones puede realizar, y, por ello debería poder desarrollar un ser vivo más complejo, lo cierto es que en muchos casos la redundancia en estas especies con un gran número de genes es muy elevada, lo cual es una estrategia evolutiva también para asegurar la supervivencia. Existen muchas estrategias adicionales, que han ido apareciendo a lo largo de la evolución, que aumentan muchísimo la complejidad estructural y funcional de un genoma, con independencia del número original de genes con el que contaran. No podemos asumir que con 20.000 genes codificantes fabricaremos 20.000 proteínas. En realidad somos capaces de fabricar muchísimas más.
Al proceso mediante el cual la información genética del ADN se convierte en ARN se llama transcripción. Al proceso mediante el cual la información genética del ARN se utiliza para fabricar la proteína que está codificada se llama traducción.
A continuación menciono tres de estas estrategias (procesamiento diferencial de los ARNs [en inglés, splicing], expresión diferencial debida a la existencia de múltiples elementos reguladores y procesamiento postraduccional de las proteínas) que resultan en un mayor número de proteínas distintas, o proteínas funcionando en células distintas en diferentes momentos de la vida del organismo. Todo ello multiplica enormemente el número inicial de proteínas que pensaríamos podrían estar codificadas por los genes de un genoma, cuyo número original carece ya de todo sentido. Además, para mayor complejidad, estas tres estrategias coexisten, lo cual da lugar a un número enorme de posibles proteínas capaces de realizar funciones ligeramente o substancialmente diferentes.
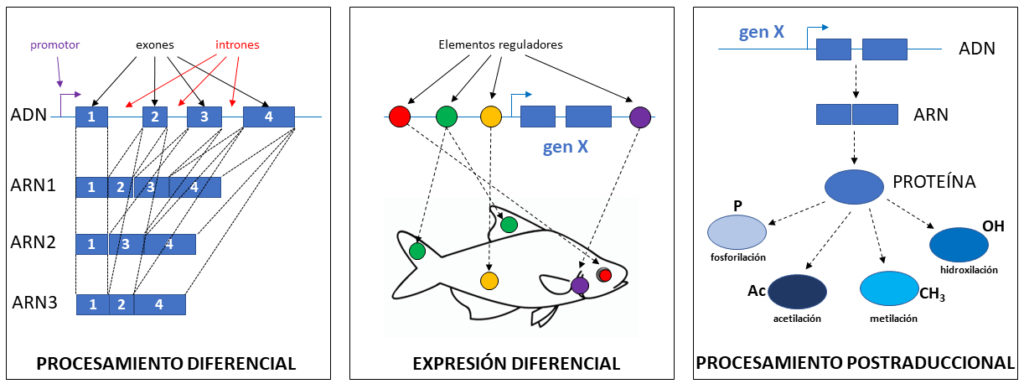
- Procesamiento diferencial: Un mismo gen puede transcribirse de diferentes maneras, mediante procesamiento diferencial, eliminado todos los intrones en orden o saltándose algún exón lo cual permite aumentar la combinatoria de las unidades (exones) que van a formar ARN distintos que, al traducirse, darán lugar a proteínas también distintas.
- Expresión diferencial: Un mismo gen puede funcionar (expresarse) en diferentes órganos del animal, o en diferentes momentos del desarrollo o del ciclo vital del animal, lo cual multiplica las funciones de una misma proteína según aparezca en un tipo celular o en otro, o según el momento en el que se exprese el gen, gracias a la presencia de múltiples elementos reguladores.
- Procesamiento postraduccional: Un mismo gen (ADN) puede transcribirse a un ARN y este dar lugar a una proteína. Ahora bien, esta misma proteína puede sufrir diversos procesamientos postraduccionales, tales como fosforilación, acetilación, metilación, hidroxilación, y muchos otros, que van a alterar la función de la proteína dando lugar a proteínas ligeramente distintas que, a pesar de estar codificadas por un mismo gen han acabado actuando en múltiples tareas.


Muy interesante saber que el genoma humano tiene alrededor de 20000 genes sin embargo, la complejidad es enorme. Gracias por el artículo
Gracias Rodrigo por tu comentario
Hola Profesor como esta?
muy bueno el articulo
conoce el blog https://sandwalk.blogspot.com/ del profesor Moran? el discute la cantidad de genes y el adn basura esta de acuerdo con el?
Una pregunta mas compleja si conocemos la estructura 3d de todas las proteinas humanas eso no permite entender su funcionamiento? ya que siempre leo proteinas que ya se conoce su estructura 3d y se descubre una nueva funcion.
Cree usted que en estas decadas podamos entender todas las moleculas (proteinas carbohidratos lipidos etc )de una celula y su funcionamiento? o del ser humano?
Estupendo compendio de información, Lluis. Gracias
Gracias Isabel, un abrazo!
Moltes gràcies LLuís. Sóc professor de batxillerat i molt sovint els teus articles són motiu de treball i reflexió a les classes.
Muchas gracias LLuís. Soy profesor de bachillerato y muy a menudo tus artículos son motivo de trabajo y reflexión a las clases.
Molt agraït Francesc! Gracias!
Está fenomenal, Lluís. Desde que entendemos ahora por el concepto de gen (tomo ya la definición para mis clases), la complejidad del genoma en relación con el ADN, todos los datos de número de genes de distintas especies (esto siempre es muy preguntado por mis alumnos) hasta lo último de procesamiento diferencial (splicing), que aunque mencionado, no viene muy bien explicado en los libros de texto. (en su día, Guillermo Peris, ya me aclaró alguna duda)
Mañana comienzo Ac. Nucléicos en 2º de bachillerato y esto junto con el anterior artículo me viene muy requetebién. Así que muchas gracias, Lluís.
Muchas gracias Marisa!
Impresionante artículo! Muchísimas gracias! Creo que he comprendido un poco mejor como funcionan los genes.
Excelente artículo. Muy buena explicación
Querido Lluís:
Repasando tu artículo, he visto que el párrafo que sigue a a la figura de los comosomas dice que el cromosoma Y tiene 108 genes cuando ese es el número de genes no codificantes. Supongo que te refieres a los codificantes que son 64.
Gracias de nuevo por este estupendo compendio.
Un abrazo
Muchísimas gracias Isabel. Te agradezco la corrección. Ya está corregido en el texto. Abrazos!
Hola Lluís. Además de la información genética de padres, sería posible que los genes contuvieran información tipo imágenes o situaciones que marquen a los individuos? Y estas pasen a los hijos?
El genoma codifica información que se transforma en ARN o proteínas, que son las que realizan las acciones. En bacterias, usando las herramientas CRISPR, se ha logrado codificar información de imagen de cinco fotogramas de una película. Lo hizo George Church en 2017. Esta es la publicación: https://pubmed.ncbi.nlm.nih.gov/28700573/
Excelente artículo, muy interesante!! a usarlo en las clases de Biología!!
Gracias Fernanda!
Leí el artículo varias veces, para poder comprender al menos un poco de lo que dice. No soy estudiante, tengo ya 61 años de edad, y por ende, la lectura de este artículo, puso algunas dudas en mi mente, no obstante, me parece interesante. Me hubiese encantado que en mi juventud, pudiera estudiar mas acerca de este tema, me gusto, me fascino y aprendí mucho. Gracias por compartirlo
Jilcamar, mantener vivo el asombro ante la vida y sus fenómenos es una actitud que enriquece la existencia.Interesante texto.
Lo felicito, excelente artículo.
Muchas gracias por la puesta al dia y la clara explicación.
Me ha gustado la nueva definición de gen y la comparativa entre especies. Es totalmente chocante que el trigo este a la cabeza de todo. ¿Cual es la especie con menor genoma?
Supongo que hay muchas por debajo del nº 100 (Streptococcus pyogenes A20) con 1.828
Un saludo
Leo este magnífico artículo unas horas antes de explicar a mis alumnos de bachillerato el concepto de gen. Qué gran actualización. Muchísimas gracias, Lluis Montoliu.
Gracias a ti por aprovechar estos artículos en tus clases.
Excelente información, aclara la cantidad de genes de nuestro genoma, ¿sólo los genes de referencia se expresan?
El ciervo Muntiacus Reevesi tiene 23 pares de cromosomas, igual que los humanos.Pero segun he leido, la cantidad de pares de cromosomas determinan la especie. Mi pregunta es: ?Por que si este ciervo tiene 23 pares de cromosomas, no es humano? ?Realmente la cantidad de pares de cromosomas no determina la especie?
El número de cromosomas NO determina la especie. Dos especies no relacionadas pueden tener el mismo número de cromosomas. Lo que determina una especie es su genoma, es la secuencia de su ADN, sus genes.
Un excelente artículo Lluís. Está muy bien explicado para que se pueda entender a la perfección. Tengo un blog sobre biología, que explico muchas cosas sobre genética, y sé lo laborioso que es a veces poner en palabras lo que se sabe para que todos lo puedan entender.
Muchas gracias Susana!
Muchas gracias Lluis por explicar tan didacticemente un tema que puede ser complejo. Graciassss.
Excelente blog!
Me encanta leerlo <3
Es como una buena rebanada de pastel