
Hace unos días ha saltado la noticia de que la empresa 23andme, una de las que se ha hecho famosa por popularizar los llamados test genéticos directos al consumidor, ha empezado a despedir trabajadores. Al parecer unos 100, un 14% de su plantilla. Esta empresa, en la que participa Google, explica, a través de su CEO Anne Wojcicki que habían empezado a descender sus ventas, tras mucho tiempo de crecimiento sostenido, casi exponencial. y tras alcanzar más de 10 millones de clientes que habían decidido obtener información genética sobre sus ancestros, sus familiares desconocidos y sobre las variantes genéticas que portaban en su genoma y su posible relación con enfermedades. ¿Qué puede haber pasado para que los clientes potenciales parezcan perder algo de interés en los test genéticos ofertados por esta empresa, cuyo nombre nos recuerda al número de cromosomas que tenemos los seres humanos: 23 ? Este descenso en las ventas no parece ser exclusivo de 23andme. Otras empresas del sector, que ofertan test y servicios similares, como Ancestry, también reportaron recientemente descensos en sus ventas. En este artículo (y los siguientes que iré publicando sobre este tema en este mismo blog) intentaré responder a esta pregunta, partiendo de mi propia experiencia como cliente de 23andme, tras haber enviado un tubito con mi saliva para que extrajeran el ADN de mis células de la mucosa bucal y obtuvieran información genética de mis ancestros, mis familiares y mis riesgos de padecer alguna de las enfermedades que esta empresa evalúa.
Esta empresa y otras similares del sector han aparecido de forma recurrente en titulares de prensa, por diversos motivos. Probablemente muchos de vosotros hayáis leído noticias en las que se anuncia que los datos agregados (no los datos de cada uno de los clientes, por separado) de los clientes de 23andme han sido vendidos a grandes empresas farmacéuticas, que esperan poder usarlos para investigar nuevas dianas y nuevos tratamientos. Estos datos también les serán útiles para decidir en qué países determinados medicamentos pueden ser más o menos efectivos, en función de la presencia relativa de variantes genéticas asociadas con una mejor o peor eficacia de determinados fármacos. No debería sorprendernos. El acceso a estos test genéticos es sencillo y relativamente barato. Y, ya sabemos que, cuando algo en la actualidad es barato o gratuito en realidad somos nosotros, nuestras variantes genéticas, quienes aportamos el valor añadido al producto (de la misma forma que lo hacemos cuando usamos aplicaciones «gratuitas» de internet en redes sociales como twitter, facebook, whatsapp, instagram. Nuestro comportamiento en estas redes sociales es la información que le aportamos a la empresa). Nosotros somos la información. Y eso lo tenemos que tener claro. La posibilidad de ceder datos agregados a terceros está incluida en las condiciones de servicio y criterios de privacidad que aceptamos al participar en los análisis. Dentro de la aplicación también podemos decidir si aportamos nuestros datos individuales, o no, anonimizados, para investigación.
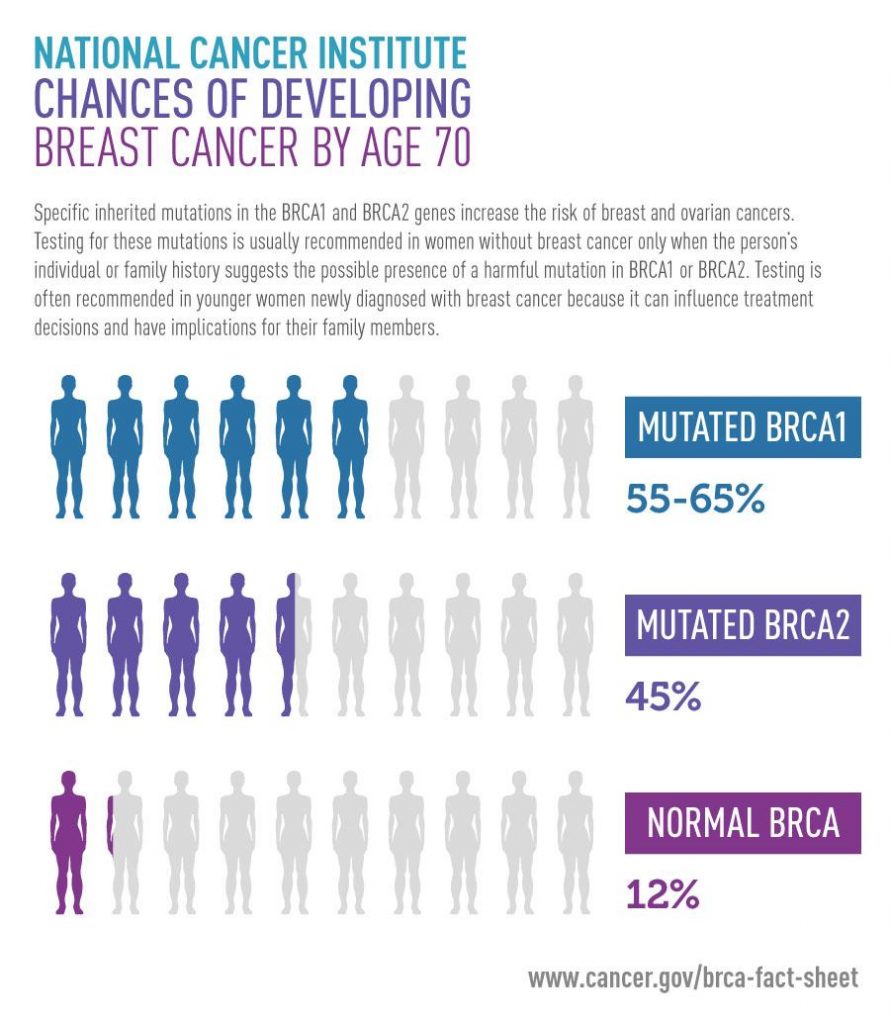
También esta y otras empresas similares han tenido problemas con la FDA, la poderosa agencia norteamericana que protege la salud pública y regula la seguridad de alimentos y eficacia de medicamentos, entre otras funciones. Particularmente en lo que respecta a los servicios que proporcionan estas empresas que podrían ser considerados «médicos» como son los «diagnósticos genéticos», y, que por lo tanto deberían ser autorizados por la propia FDA. Por eso estas empresas se encargan de recordar en toda su documentación que la información que facilitan no debe ser considerada un diagnóstico genético, sino solamente a titulo informativo, educativo o de investigación. En España, hay que recordar que un diagnóstico genético debe ser solicitado por un médico experto en genética, que juzgará la necesidad y oportunidad de encargarlo, y ser recibido por un médico o profesional sanitario experto en genética, que se encargará de explicar al paciente, en lo que conocemos como consejo genético, los resultados obtenidos, así como los riesgos, relevancia y trascendencia familiar que conllevan. Por ello, hay que ser conscientes de los riesgos que pueden generar estos test genéticos directos al consumidor, las posibles alarmas y la desinformación que pueden producir al no pasar en ningún momento por las manos de ningún médico o profesional sanitario. La presencia de una determinada variante puede estar asociada a una determinada probabilidad (no certeza) de desarrollar una patología. Y este tipo de situaciones inesperadas, a pesar de todos los avisos de estas empresas, ya ha empezado a suceder con personas que descubren que son portadoras de mutaciones con una alta probabilidad (que no certeza) de desarrollar un determinado tipo de cáncer. Guillermo Peris se refería recientemente también a los problemas éticos asociados a descubrir la presencia de mutaciones en menores, sin trascendencia clínica, que, sin embargo, su efecto sí acabará manifestándose en la etapa adulta.
Finalmente, han aparecido noticias en las que se explica que el FBI ha usado algunas de estas empresas para encontrar familiares de criminales no localizados y así, a través de ellos, poder detener a los asesinos y resolver crímenes que llevaban muchos años sin poder cerrarse. Algunas empresas abiertamente afirman haber proporcionado estos datos/accesos al FBI, y otras, como 23andme indican que resistirán (pero, lógicamente no podrán impedir) el acceso de autoridades policiales a sus bases de datos, ante un mandato judicial. Esto también hay que tenerlo en cuenta.

Probablemente todas estas noticias, y algunas más que han surgido sobre las prácticas de estas empresas de test genéticos directos al consumidor y los riesgos que asumen los clientes, sobre el mantenimiento de su privacidad, han acabado por asustar o, por lo menos, hacer recapacitar a futuros clientes que ahora quizás mediten un poco más si acceden a participar en esta operación, en la que claramente quien obtiene mayor información y posibles beneficios son las empresas, no los participantes. Esto podría explicar, en parte, el descenso en las ventas de kits de participación.
Sin embargo yo creo que, además, debe considerarse la información real de nuestros genomas que obtenemos de estas empresas. ¿Qué tipo de información obtenemos? ¿Vale la pena? ¿Cómo de fiable es? ¿Qué relevancia tienen los resultados que obtengo? Y para ello, como científico, me propuse, como muchos otros investigadores antes que yo, realizar el experimento en primera persona, y solicitar los servicios de una de estas empresas.
Empecemos por el principio
Hace algún tiempo un colega y buen amigo, genetista como yo, me habló de la empresa 23andme, y que toda su familia (él, su mujer y sus tres hijos) se habían hecho los test y estaban entretenidos y satisfechos con los resultados que habían obtenido, lo cual les había permitido, en su caso (es una familia americana pero de múltiples ancestros europeos) conocer algo más el origen de sus antepasados. No pedían más, y habían quedado contentos. Me pareció curioso y sorprendente la información que habían obtenido por un módico precio, que, desde entonces, no ha hecho más que reducirse, hasta bajar de la barrera de los $100 USD. Algún tiempo después nos llamó un día nuestra hija, que también es genetista y trabaja en Suecia, donde está terminando su tesis doctoral sobre genética evolutiva de hongos, para decirnos que «estábamos bien, que no teníamos que preocuparnos», ni mi mujer ni yo. ¡Anda! ¿Cuál era el motivo de tal inesperada buena nueva? Lo habréis ya adivinado. Efectivamente, como miembro de una generación de jóvenes 2.0, crecidos en la cultura de compartir información, y en el uso habitual de las nuevas tecnologías, ella también había analizado su genoma con 23andme. Y, claro, su genoma es producto, al 50%, del que hereda de su madre y del mío. Cada uno de nosotros dos le pasamos un 50% de nuestro genoma para constituir su nuevo genoma. Y, revisando las variantes genéticas potencialmente asociadas a enfermedades graves no parecía ella tener ninguna, luego tampoco las tendríamos nosotros. Al menos en el 50% que le habíamos transmitido.
Con mi mentalidad bioética enseguida pensé que esto era una verdadera revolución, hasta cierto punto increíble, que nos había explotado delante de todos y en la que apenas habíamos tenido tiempo de reaccionar ni de analizar sus consecuencias. Mi hija había podido obtener información de su genoma al optar, voluntariamente, a usar esta empresa para analizar su genoma. Nada que objetar en este aspecto. Era su decisión como persona adulta y libre. Pero, simultáneamente, esto nos implicaba a nosotros dos, sus padres (y a su hermano). A ninguno de los tres nadie nos había preguntado si queríamos o no saber si «estábamos bien» o no. Si queríamos saber si eramos portadores o no de variantes potencialmente asociadas a patologías. Inmediatamente me di cuenta que era una batalla perdida. Estos test genéticos directos al consumidor habían llegado para quedarse y en poco tiempo se convirtieron en tendencia y en algo extraordinariamente popular. Ese año, al llegar las navidades, nuestra hija nos regaló (como hicieron centenares de miles de personas en todo el mundo) un test genético de 23andme para cada uno de nosotros tres, y decidimos experimentar, en primera persona, qué información podríamos obtener de 23andme.
La tecnología que usa 23andme
La empresa 23andme no secuencia el genoma completo de sus clientes. Lo que hace es analizar una serie de variantes genéticas que tenemos todos en nuestros genomas y que nos diferencian. En general, dos personas cualquiera nos parecemos a nivel genómico al 99,9%. Esto quiere decir que nos diferenciamos en apenas un 0,1%. Si nuestro genoma tiene tres mil millones de nucleótidos (en realidad tres mil millones de pares de bases: G, A, T y C, dado que el ADN es una molécula bicatenaria) esto nos dice que nos diferenciamos en, por lo menos, unos 3 millones de posiciones. En realidad las variantes conocidas experimentalmente, tras analizar muchos genomas, son bastantes más, alrededor de 4 a 5 millones de posiciones en nuestro genoma. A estas variantes genéticas de una letra (de un nucleótido) que pueden ser distintas en una determinada posición se les llama SNP (del inglés Single Nucleotide Polymorphism). Algunas de estas variantes, también llamadas polimorfismos, están asociadas a patologías o, mejor dicho, al riesgo de padecerlas, lo cual no es exactamente lo mismo.

Desde sus inicios como empresa de venta de test genéticos directos al consumidor 23andme ha estado usando diferentes chips de ADN con un número creciente de polimorfismos. Uno de los últimos chips que usa 23andme se llama V5 y es capaz de leer y detectar unos 650,000 polimorfismos a lo largo de todo el genoma, cuya distribución cubre, más o menos, gran parte de las regiones de los 23 cromosomas, aunque con algunos espacios en blanco no representados. En otras palabras, lo que 23andme lee, en principio, de nuestro genoma no es el genoma completo (las 3.000 millones de letras), ni tan siquiera la totalidad de secuencias genéticas que codifican los aproximadamente 20.000 genes que tenemos (lo que llamamos exoma completo, que representaría aproximadamente un 2% del genoma, teóricamente unas 60.000.000 de letras), sino que tan solo lee unas 650.000 de ellas, lo cual representa unas 100 veces menos, alrededor del 0,02% de nuestro genoma. Haríamos bien en recordar esta cifra, puesto que todo lo que viene a continuación, toda la información que obtenemos de 23andme deriva de esta cantidad casi insignificante de nuestro genoma. Evidentemente estos polimorfismos están seleccionados e incluyen la mayoría de los que han sido asociados con características genéticas determinadas o con la posibilidad de desarrollar o padecer una determinada patología.
Los resultados de 23andme
A los pocos días de remitir las muestras de saliva recibimos notificación de que habían recibido los tubos en la empresa, y unas cinco semanas después ya nos informaron que teníamos los resultados listos para ser consultados. ¿Qué tipo de resultados son los que muestra 23andme? Son de varios tipos. Según su clasificación las categorías principales son: ancestros, salud, características genéticas (p.e. color de los ojos) y familiares. En este y en los siguientes artículos de esta serie me iré refiriendo a cada uno de estos apartados. Para empezar, en este primer artículo, me referiré solamente a los datos de mis ancestros, mis orígenes. Quiero señalar que, antes de poder acceder a los resultados, hay que revisar unos tutoriales en los que la empresa explica la tecnología que aplica, la información que puede obtenerse y sus limitaciones. Hay vídeos en YouTube también en los que usuarios de 23andme describen, con todo lujo de detalles, sus resultados y cómo interpretarlos.

Mis ancestros
Probablemente en Europa tenga menos interés preguntarse cuáles son tus orígenes, cuando ya sabes (como es mi caso) que tus cuatro abuelos nacieron en una misma región de un mismo país. Y cuando conoces o sospechas que muy probablemente sus progenitores y antepasados también anduvieron mayoritariamente por las mismas tierras. Pero puedo entender que esto tenga un mayor interés en países de «reciente» creación, como EE. UU., en los cuales diversas olas de inmigrantes, llegados de muchos orígenes distintos, han dado lugar a una población mezclada (el famoso «melting pot» norteamericano) en la que sus habitantes, seguramente mucho más diversos que en Europa, desean saber quiénes fueron sus antepasados. Esto explica el gran éxito que han tenido estas empresas de test genéticos, de genealogía y de ancestros en ese país. Y estos son los primeros resultados que te ofrece 23andme con tu ADN, tras analizar los 650.000 SNPs de tu genoma. ¿Cuáles son mis ancestros? Pues, según 23andme, son estos:

Según los análisis realizados por 23andme yo soy europeo al 99,9%. Apenas han encontrado un 0.1% de las variantes analizadas (unas 650 de las 650.000 testadas) que no parecen encajar en su base de datos de variantes europeas. Evidentemente no me sorprende. Hasta donde recordamos en la familia todos mis antepasados han sido europeos. Bueno, primera confirmación obtenida que no conlleva ninguna sorpresa. Seguidamente veo que tengo un 58,2% de europeo del Sur. Lógico también. Y, además, son capaces de determinar que en un 48,6% de las variantes testadas (casi la mitad de ellas) derivan de un origen hispano-portugués, en particular Cataluña. ¡Bingo! Yo nací en Cataluña, como mis padres, como tres de mis cuatro abuelos, como muy probablemente la mayoría de mis ocho bisabuelos. También parece que tengo un poco de origen italiano (un mísero 1,7%), nada descartable, especialmente en aquellos que como yo (y la mayor parte de mi familia) hemos nacido en la costa mediterránea, visitada históricamente por muchos marinos… también italianos. Se añade un 7,9% indefinido pero sureño europeo. Pues muy bien. Soy europeo y del sur. Ya lo sabía, pero en fin, está bien confirmarlo.
El 38,3% de mis variantes parecen apuntar a un origen europeo central, del noroeste, una zona indefinida que incluye la Europa central, con Francia y Alemania, y las islas británicas e irlandesas. De hecho el análisis detecta un 17,9% de origen francés o alemán (hasta cierto punto también lógico, viniendo de Cataluña, una tierra fronteriza que ha sido visitada por franceses y centroeuropeos en multitud de ocasiones a lo largo de la historia. Y un 7,9% de británico e irlandés, lo cual tampoco me sorprende, pues mi abuelo paterno, nacido en Aragón, en la franja fronteriza con Cataluña, era pelirrojo, lo cual sugería que algunos de sus antepasados pudieron provenir de Inglaterra, Gales, Escocia o Irlanda, donde existen muchos más pelirrojos que en el resto de Europa. Se completa el porcentaje europeo del noroeste con otro valor indefinido, del 12,5% que también viene de esa zona, sin mayor concreción. Lo mismo ocurre con el 3,4% de mis variantes polimórficas, que tienen origen europeo, pero sin poder concretarse más.
¿Alguna sorpresa? Pues la verdad es que no. Los resultado parecen confirmar lo que ya sabíamos o sospechábamos en la familia. Quizás la única fuente indeterminada que puede haber dado algo de «salsa» a estos análisis es uno de mis bisabuelos, a quien le debo mi curioso segundo apellido, materno (que es un nombre de pila en realidad), que fue abandonado o donado a un orfanato, y del cual, lógicamente no sabemos su origen. Ese puede ser el que aportó los orígenes menos esperados. Pero eso es pura especulación.
Preguntémonos ahora cómo han obtenido estos porcentajes en 23andme. ¿Cual es la ciencia detrás de los análisis de ancestros? El análisis de ancestros en 23adme es relativamente complejo e implica diversos procesamientos de los datos crudos y análisis estadísticos. De nuevo hay que recordar que los chips de SNPs solamente dicen qué letra hay en una determinada posición. Pero dado que hay dos letras en esa posición (un nucleótido y su complementario en la cadena contraria, uno heredado de nuestro padre y el otro heredado de nuestra madre) no sabemos de quién hemos heredado qué letra. Pero el análisis de grupos de letras que (habitualmente) están cercanas en el genoma (tras analizar centenares de miles de genomas) les permite aseverar, con una cierta probabilidad, que determinadas letras corresponden a un mismo cromosoma, porque «suelen» ir juntas en una determinada población. Al conjunto de letras (SNPs) que se heredan conjuntamente, a ese fragmento del cromosoma que viaja unido y se hereda unido de padres a hijos lo llamamos haplotipo. Cuando un conjunto de haplotipos son característicos e identifican una población se llama haplogrupo. Estos haplotipos pueden ser característicos de determinadas poblaciones en el mundo. Determinadas agrupaciones de SNPs pueden ser características de genomas europeos del sur, o centroafricanos, o chinos o japoneses. Y esa es la información, con un determinado nivel de confianza (por defecto del 50%, que es altamente especulativo), que analiza y traslada 23andme. Si variamos ese nivel de confianza tendremos resultados más conservadores, más «creíbles», menos especulativos, pero, lógicamente también, mucho menos definidos. 23andme analiza actualmente 216 poblaciones mundiales.
Los datos anteriores de mis ancestros están calculados con un 50% de nivel de confianza. Si ahora los recalculo subiendo al 90% ese parámetro los resultados que obtengo de mis ancestros son:
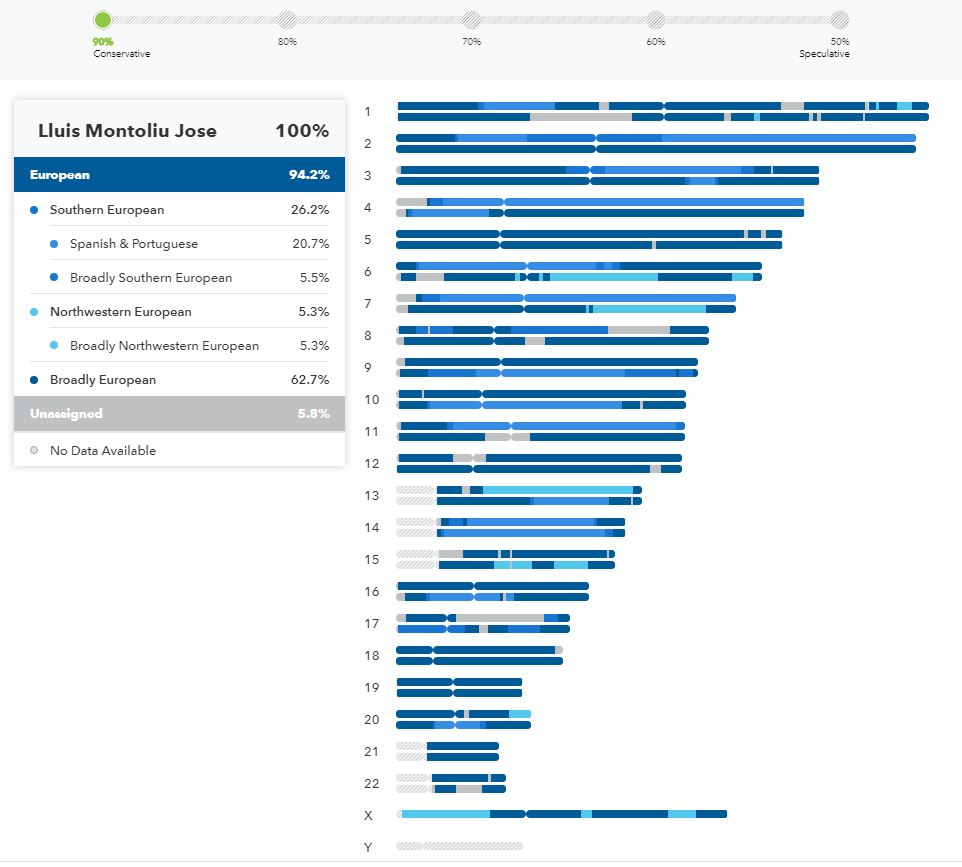
Si consultamos los porcentajes (ahora calculados al 90% de nivel de confianza) observaremos que sigo siendo mayoritariamente europeo, pero algo menos. En lugar del 99,9% que obtenía con el 50% de nivel de confianza ahora obtengo un nada desdeñable 94,2%, y paso de no poder asignar el 0.1% a no saber asignar el 5,8% de las variantes analizadas. Dentro de Europa ahora mi origen sureño baja estrepitosamente al 26,2% (más de la mitad que el valor anteriormente observado), aunque, dentro de este porcentaje, sigo siendo mayoritariamente hispano-portugués (20,7%). El porcentaje de genoma que deriva de ancestros de la Europa Central o del Oeste también cae al 5,3% (de un 38,3% anterior). Y, al ser mucho más conservador con este intervalo de confianza, sigo sin poder asignar más allá de variantes (haplotipos) «ampliamente europeos» nada menos que un 62,7% de mis variantes. Estos resultados, variables según el nivel de confianza que especifiquemos, nos recuerdan la cautela que tenemos que tener con todos estos análisis y la distancia y prudencia que tenemos que tomar antes de aseverar que derivamos de italianos, irlandeses, griegos o polacos, lo cual me temo, es uno de los peligros que tiene la interpretación directa de los resultados que presenta 23andme por defecto (al 50% de confianza). Sinceramente, creo que estamos lejos, con estos resultados de 23andme, de poder determinar, con exactitud, el origen de nuestros antepasados. Sería necesario secuenciar una mayor parte del genoma y consultar otro tipo de bases de datos, de genomas completos, con más referencias, para poder aseverar con mayor certeza el origen de nuestros ancestros.
23andme usa como referencias para las poblaciones los datos de haplotipos y haplogrupos accesibles en bases de datos internacionales, pero también los orígenes de sus propios clientes, exactamente de 11.744 clientes, de origen verificado (frente a 2.663 referencias poblacionales obtenidas de bases de datos públicas). La empresa indica que a medida que su base de datos con genomas de referencia de diferentes orígenes vayan aumentando su análisis de ancestros será progresivamente más fiable. Evidentemente esto tiene el peligro de una representación sesgada, no proporcional, de las diferentes poblaciones. Por ejemplo, si hay pocos clientes de origen africano entonces las referencias de genomas africanos estarían infrarepresentadas y las estimaciones de ancestros tenderían a ser subóptimas. Y esto es particularmente cierto en África, cuando acabamos de conocer que existe aproximadamente un 10% de secuencias genéticas del genoma humano que solo se encuentran en África y que desconocíamos. Lo cuenta estupendamente Gemma Marfany en su artículo semana en El Nacional.
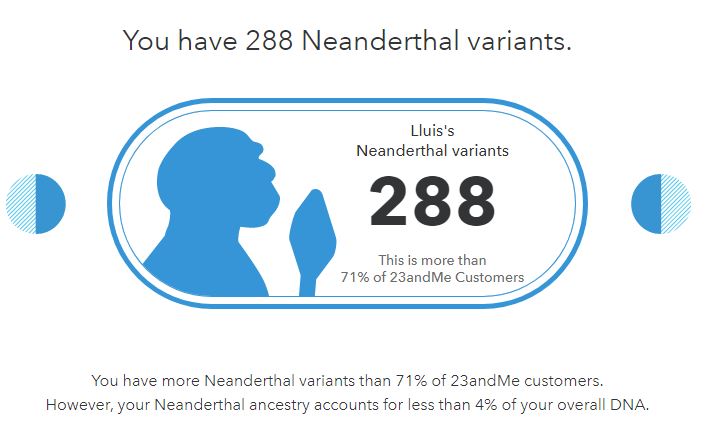
Seguidamente el análisis de ancestros se completa con el análisis del porcentaje de ADN derivado de Neandertal que tengo en mi genoma. Los Neandertales se extinguieron hace unos 40.000 años pero hay evidencias que indican que se cruzaron con Homo sapiens probablemente varias veces. El análisis de 23andme me dice que llevo 288 variantes (polimorfismos) de origen Neandertal, de las 1436 variantes que analizan, dispersas por todo el genoma. 252 de todas ellas las tengo en uno de los dos alelos, y en 18 ocasiones las tengo en los dos alelos. Y que esto me sitúa por encima del 71% de los clientes de la empresa, a pesar de que el contenido Neandertal de mi genoma es inferior al 4%. Pues estupendo, ¿soy más prehistórico que los demás? ¿más bruto? No tengo yo sensación de que este conocimiento vaya a cambiar mi vida, pero bienvenido sea.
Una de las variantes Neandertal que llevo, en heterocigosis (solamente en una de las dos copias del SNP, heredada o bien de mi padre o de mi madre) es la variante rs7544462, una A por una C, en el gen MEAF6, que parece determinar que sea un 1% más bajo de estatura. Si tuviera dos Cs en esa posición, las dos de origen Neandertal, todavía tendría la posibilidad de ser algo más bajo que la media. Evidentemente la altura que consigue un individuo no solamente es producto de su genética (de múltiples genes y sus productos codificados) sino también del entorno, de su alimentación y del tipo de actividad física que realice.
Finalmente los datos de ancestros incluyen el haplogrupo materno al que pertenezco, analizado mediante el estudio del ADN mitocondrial, que se hereda solo por línea materna y que yo heredé de mi madre, y ella de mi abuela, y esta de mi bisabuela, etc… transmitido a través de las mujeres de mi familia. Los análisis me dicen que pertenezco al haplogrupo materno C4a1, al parecer relativamente poco común entre los clientes de 23andme. Al parecer mi componente materno (mitocondrial) deriva de una mujer que vivió hace unos 11.500 años. Estupendo.
Para el análisis del haplogrupo paterno 23andme analiza las variantes del cromosoma Y, que de forma complementaria a lo que sucede al ADN mitocondrial por via materna, solo se transmite de padres varones a hijos varones, prácticamente sin recombinación. Al parecer pertenezco al haplogrupo paterno R-CTS1211, que deriva de un hombre que vivió hace unos 6.000 años. Estupendo también. Tanto para el haplogrupo materno como el haplogrupo paterno 23andme me ilustra la pertenencia a los grupos indicados con personajes o momias históricas encontradas en determinados lugares del mundo, con quienes comparto el ADN mitocondrial y el cromosoma Y. Estupendo.
En el fondo, como dice Juan Ignacio Pérez Iglesias, el número de ascendientes es muy inferior al que podemos calcular teóricamente, dado que muchos de nuestros antepasados son lógicamente compartidos. Se estima que todos los seres humanos actuales compartimos un antepasado común que vivió hace unos 3.400 años.
Y hasta aquí el primero de los artículos sobre 23andme comentando mis resultados de ancestros. Próximamente seguiré comentando los datos de salud, de características genéticas y sobre mis familiares. Permaneced atentos.
NOTA: No tengo ninguna relación comercial con las empresas a las que me refiero en este artículo, más allá de ser uno de los más de 10.000.000 de clientes que ha utilizado sus servicios para obtener información genética sobre su genoma. En este artículo ni aconsejo ni desaconsejo usar estas empresas, simplemente expongo mi opinión a partir de los resultados obtenidos y en función de otros comentarios y artículos publicados sobre este tema. Cada persona es por supuesto libre de decidir si quiere usar los servicios de estas empresas o no, tras leer este y otros muchos artículos similares que existen disponibles sobre esta temática.


Excelente !! Tu artículo me ha demostrado hasta donde se pueden considerar estos estudios, llamémosle, superficiales.
Creo que sólo como curiosidad personal.
Sin embargo para los sudamericanos no dejan de ser interesantes como curiosidad, ya que nuestros abuelos y bisabuelos son originarios de muy distintas zonas del mundo. En mi caso, todos europeos de una franja del planeta muy definida pero de distintos países.
Muy interesantes tus artículos que sigo atentamente por tweeter.
Espero lo que falta de tu informe genético.
Desde Argentina, Beatriz Molinari. Galdeano
Muchas gracias Beatriz!
Hola,
Tu artículo me pareció muy interesante, cierto es que algunas cuestiones (a la hora de entenderlo) se me escapan. Pero está todo muy bien expresado y valorado. Enhorabuena
Muchas gracias!
Muy interesante este articulo quisiera saber si es posible si una persona puede localizar algunos familiares de su padre, aún no teniendo ninguna información de él .Gracias por su tiempo Cristina!!
Hola Cristina, para localizar un familiar en 23andme o empresas similares necesitas, ante todo, proporcionar tu propio ADN al sistema. Y será entonces cuando el sistema pueda localizar a otras personas con quienes compartas algunos fragmentos de tu genoma. Si compartes alrededor del 50% del genoma probablemente se tratará de un progenitor.
Hola Lluis,
Primero de todo, muchas gracias por compartir con tod@s su experiencia. Soy una persona que no me dedico a la genética ni a la historia. Pero me gusta mucho leer sobre el tema, ¿su ADN-Y R-CTS1211 es propio de Italia?, ¿su antecesor es el R1a cual proviene de Rusia?.
Muchas gracias por todo,
Muchas gracias por su artículo, profesor Montoliu. Despeja muchas de las dudas que hay sobre este tipo de pruebas.
Sin embargo, tengo una cuestión que plantearle:
Esos 650.000 SNP se buscan en la totalidad del genoma, 6000 millones de pares de bases, pues entiendo que el genoma haploide (22 autosomas + x + y) comprende 3000 millones de pares de bases, ¿no es así?
Hola Javier, el genoma humano tiene un tamaño aproximado de 3000 millones de nucleótidos (contabilizando una de las dos cadenas del ADN), o letras, como se suele indicar, que corresponden naturalmente a 3000 millones de pares de bases, luego, si cuentas las dos cadenas de ADN serían 6000 millones de nucleótidos.
cordiales saludos
Lluis
Entiendo que cuando se refiere a «las dos cadenas de ADN», se refiere a las parejas de cromosomas homólogos (un juego heredado de nuestro padre y otro juego heredado de nuestra madre). Son pues, 6000 millones de nucleótidos. Y es que hay que tener en cuenta los dos juegos cromosómicos, pues los SNPs pueden encontrarse tanto en cromosomas heredados de nuestro padre como en los heredados de nuestra madre.
En esta entrada comenta: «En general, dos personas cualquiera nos parecemos a nivel genómico al 99,9%. Esto quiere decir que nos diferenciamos en apenas un 0,1%. Si nuestro genoma tiene tres mil millones de nucleótidos (en realidad tres mil millones de pares de bases: G, A, T y C, dado que el ADN es una molécula bicatenaria) esto nos dice que nos diferenciamos en, por lo menos, unos 3 millones de posiciones»
Si nuestro genoma tiene 6.000 millones de nucleótidos, el 01% son 6 millones de posiciones.
También comenta: «tan solo lee unas 650.000 de ellas, lo cual representa unas 100 veces menos, alrededor del 0,02% de nuestro genoma»
Si nuestro genoma tiene 6000 millones de nucelótidos, 650.000 nucleótidos suponen el 0,01083%
Estos números están de acuerdo con los datos que aporta en su entrada «El tamaño del genoma humano»
Muchas gracias
Sí, gracias, por eso me refiero en el texto a «en realidad tres mil millones de pares de bases»
Hola, muy interesante el artículo. La cuestión que planteó es sencilla e importante para mí. Mi abuela materna fue una niña expósita, nunca podré saber su procedencia por los procedimientos habituales, por lo que me hice el test en 23&me. El haplogrupo materno es T1a1, no demasiado frecuente, parece ser. Mi pregunta es: puedo confiar en que las personas con un cierto grado de parentesco (<1%) según 23&me con las que comparto el haplogrupo materno, compartan ancestros recientes? (Tatarabuelos…)
Muchísimas gracias por su respuesta