Las maravillosas herramientas CRISPR-Cas de edición genética no dejan de sorprendernos, por su tremenda versatilidad y aplicabilidad en muchos campos de la biología, siempre y cuando se trate, de alguna manera, de interactuar con el material genético, con las moléculas de ácidos nucléicos, sean estas ADN o ARN. Estas herramientas CRISPR derivan de sistemas que, con el mismo nombre, fueron descubiertos primero en bacterias y arqueas, por el microbiólogo español Francis Mojica, donde funcionan como un verdadero sistema de defensa bacteriano frente a los virus. Mojica fue quien también les puso el nombre (CRISPR, acrónimo en inglés de repeticiones cortas palindrómicas agrupadas y regularmente espaciadas) por el que todos las conocemos hoy en día. Estas mismas CRISPR han sido resaltadas por la Academia Sueca de Ciencias con el Premio Nobel 2020 de Química a dos investigadoras, Emmanuelle Charpentier y Jennifer Doudna, por proponer en junio de 2012 su uso como herramientas de edición genética. Y, desde entonces, se han usado en diversos campos: para el desarrollo de terapias génicas frente a enfermedades graves de la sangre, para tratar cánceres refractarios o enfermedades raras pediátricas sin cura. También para diagnosticar la presencia de virus o mutaciones, y hasta para detectar y atacar el coronavirus SARS-CoV-2, causante de la pandemia actual COVID-19.
Pero es que la versatilidad de las herramientas CRISPR no tiene parangón. Ni la imaginación de los miles de investigadores tampoco, quienes continuamente están desarrollando nuevas variantes y aplicaciones con las que sorprendernos, basadas directa o indirectamente en las CRISPR. Este es el caso de un artículo científico reciente (Nuñez et al. Cell, 2021) publicado a principios de este mes de abril de 2021, que es el que quiero comentar en esta nueva entrada de mi blog de gen-ética. En este trabajo se usan unas nuevas variantes CRISPR para encender (CRISPRon) y para apagar (CRISPRoff) genes a voluntad, de forma análoga (pero de forma mucho más eficiente, sistemática y elaborada) a cómo os contaba hace unos meses que podían también encenderse y apagarse genes mediante antibióticos.
En este nuevo estudio, realizado por los laboratorios de Jonathan S. Weissman y Luke A. Gilbert, los dos en la UCSF, los investigadores han modificado el sistema CRISPR-Cas9 inicial hasta convertirlo en una herramienta «interruptor», que permite encender y apagar genes a voluntad, y mantener los estados inactivos o activos durante largo tiempo en las células. La clave de estas nuevas variantes es la proteína dCas9 (del inglés dead Cas9, o Cas9 muerta). Esta es una endonucleasa Cas9 que ha sido mutada en sus dos dominios de corte de ADN ( D10A en el dominio RuvC y H840A en el dominio HNH), responsables de cortar cada una de las dos cadenas de la doble hélice del ADN y, por lo tanto, se convierte en una nucleasa incapaz de cortar ya el ADN. ¿Y para qué sirve una nucleasa que ya no puede cortar el ADN? Ahí está el truco. No puede cortar el ADN pero sigue pudiendo posicionar la dCas9 en una localización determinada del genoma, gracias a su guía de ARN que se apareará con nuestro gen de elección. Esta genialidad se le ocurrió a Stanley Qi (en aquellos momentos en UCSF, hoy en Stanford) quien junto a un grupo de investigadores en 2013 (entre los que estaban los dos mencionados, JS Weissman y LA Gilbert, y también Jennifer Doudna) presentó por vez primera la dCas9 como un sistema para interferir con la expresión génica, para bloquear la transcripción por parte de la RNA polimerasa (RNAP), y por eso lo llamaron CRISPRi (i de interferencia).
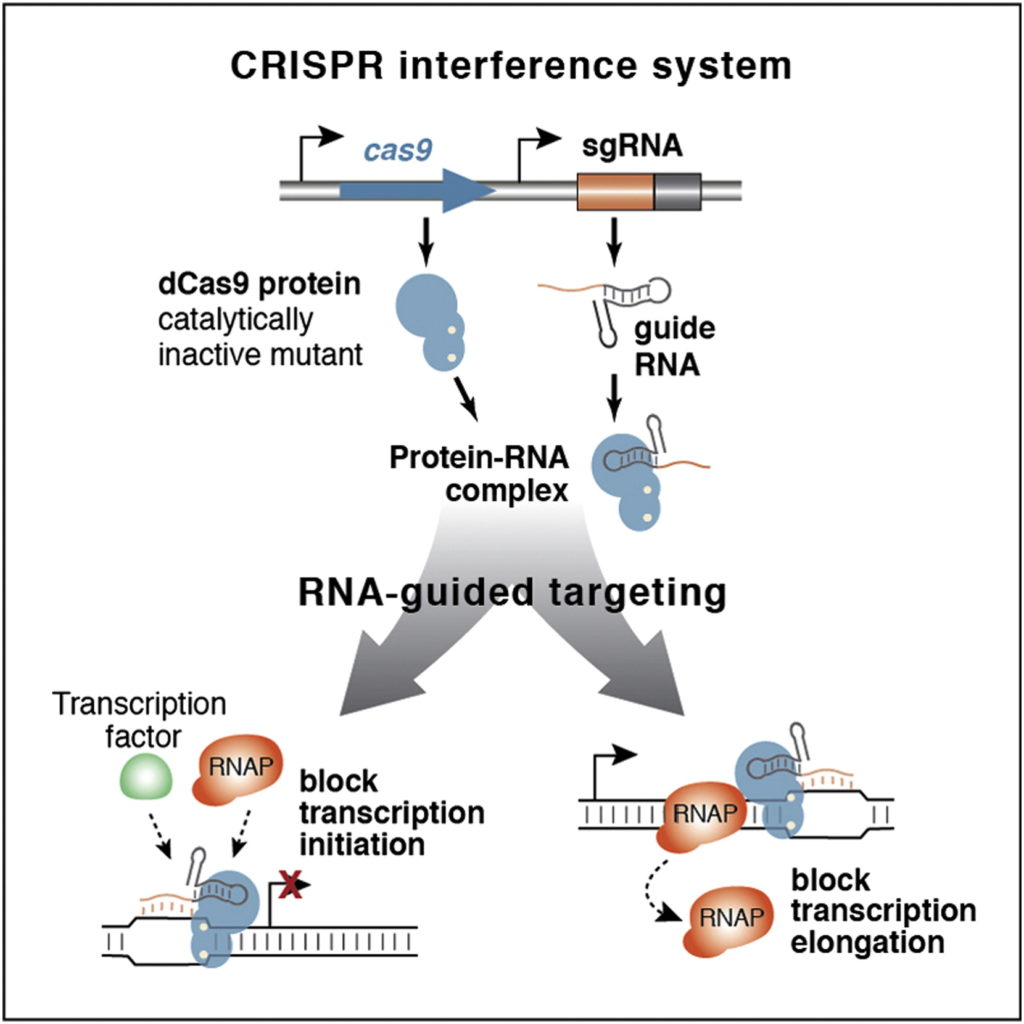
Poco después, tras aparecer la primera versión de una herramienta CRISPR-Cas9, basada en dCas9, capaz de interferir con la transcripción (CRISPRi), se constató que podía mejorarse todavía más esta interferencia si se le añadía un dominio «represor», un fragmento de otra proteína con actividad represora, capaz de inactivar la expresión génica a través de diversas estrategias (promoviendo la metilación del ADN circundante, la metilación de las histonas, ). Y entonces surgieron las primeras proteínas dCas9 de fusión, formadas por la propia dCas9 y un dominio efector que tanto podía ser promotor (activador) como represor (inactivador) de la expresión génica. En todos estos casos la dCas9 era incapaz de actuar como nucleasa pero seguía siendo capaz de interaccionar con la guía de ARN (y esta de aparearse con su secuencia complementaria del ADN o ARN) por lo que la dCas9+guía ARN era capaz de posicionarse en algún sitio concreto del genoma y, una vez allí, dejar que la actividad del dominio efector, activadora o inactivadora, realizase su función.
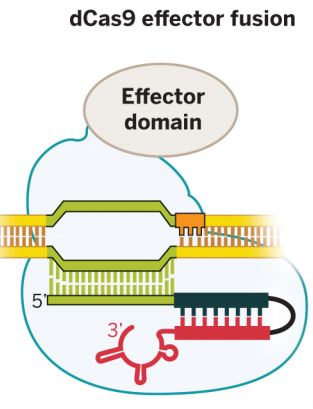
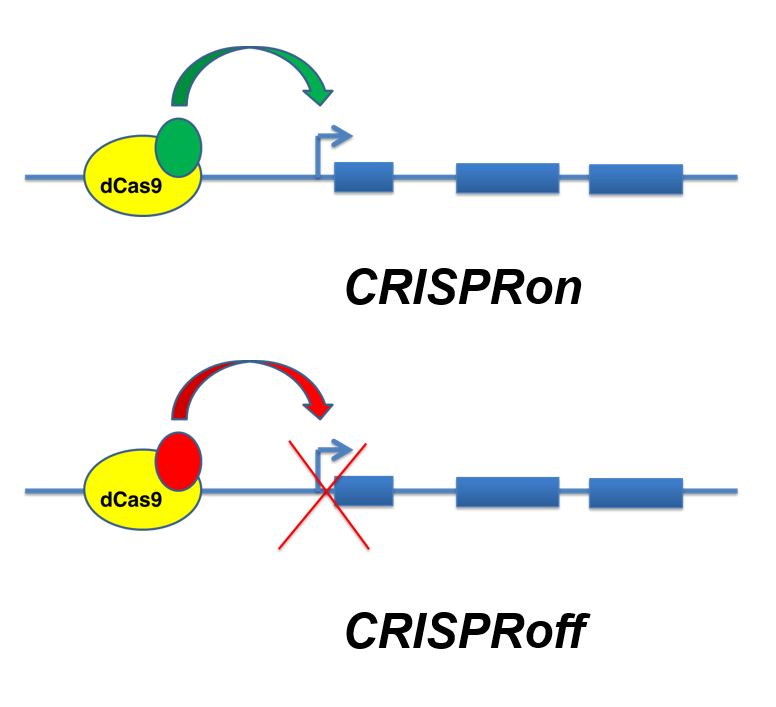
El uso de proteínas de fusión basadas en dCas9 permitió desarrollar un nuevo juego de variantes CRISPR capaces de interaccionar no ya con la capa «genética» de nuestro genoma, sino también con la capa de información «epigenética«, promoviendo la expresión de los genes (por ejemplo, a través del dominio efector activador VP64, acetilando las histonas de los nucleosomas, eliminando radicales metilo del ADN) o reprimiendo su expresión (por ejemplo, a través del dominio efector represor KRAB, o metilando la secuencia circundante de ADN). El nuevo trabajo de investigación que acaba de publicarse, con las nuevas variantes CRISPRon y CRISPRoff precisamente lo que hace es desarrollar nuevas variantes de proteínas de fusión dCas9, mucho más elaboradas, más evolucionadas, para potenciar la activación o inactivación de genes a voluntad, respectivamente.
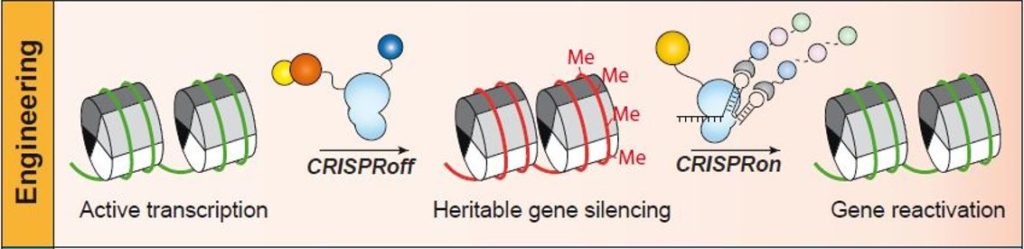
La estrategia que hay detrás de la nueva herramienta CRISPRoff es muy poderosa, pues combina la proteína dCas9 no con un dominio efector represor sino hasta con tres dominios efectores represores diferentes: KRAB y las dos metilasas Dnmt3A y Dnmt3L, capaces de inducir la hipermetilación (Me) del ADN circundante (en las citosinas de los dinucleótidos CpG) y, con ello, el silenciamiento génico, la represión estable (¡y heredable!) del estado inactivo del gen. CRISPRoff es una herramienta CRISPR, una nueva variante basada en dCas9, capaz de silenciar fuertemente los genes identificados por la guía de ARN.
De igual forma, la estrategia que hay detrás de la nueva herramienta CRISPRon es también muy poderosa, pues combina la proteína dCas9 fusionada con el dominio Tet, que tiene actividad desmetilasa (y es capaz de retirar los radicales metilo del ADN), y con ello es capaz de reactivar la expresión de un gen silenciado, potenciada con una guía de ARN a la que se le añaden unos lazos extra MS2, que se unen específicamente a la proteína MCP, que, a su vez, puede unirse hasta con tres dominios efectores de activación distintos: VP64, p65-AD o Rta, en diferentes combinaciones. Y esa reactivación por CRISPRon permite hacer reversible la inactivación previa por CRISPRoff.
Las aportaciones técnicas de este trabajo Nuñez et al. Cell (2021) van a tener un impacto enorme en biología, en nuestra capacidad de interactuar epigenéticamente sobre los genes, promoviendo su silenciamiento específico con CRISPRoff y reactivando su funcionamiento con CRISPRon. El silenciamiento es estable, muy eficaz (80 al 90%) y se transmite a través de las divisiones celulares durante muchos días después de haber desaparecido la herramienta CRISPRoff, o tras producirse la diferenciación celular, por ejemplo desde unas células pluripotentes a otras más especializadas. Incluso sobre aquellos genes que no tienen secuencias próximas ricas en dinucleótidos CpG, que son la diana para ser hipermetilados.
Muchos investigadores esperan que los reactivos para poder utilizar estas nuevas variantes CRISPR sean puestos libremente a disposición de la comunidad científica a través del repositorio Addgene, tal y como han anunciado los autores del trabajo donde se describen las herramientas CRISPRoff y CRISPRon.


¿Y esto no sale en loa telediarios? ¡¡QUE PASADA!!!
Efectivamente Óscar, hay muchas noticias sorprendentes, de gran potencial e impacto, que no llegan a la sociedad pero que sin embargo creo que merece la pena comentarlas. Humildemente este blog intenta ir solucionando estas carencias comentando temas que creo deben ser conocidos por toda la sociedad. Gracias.
Sería muy bueno que lo.pusieran a disposición de los científicos para ayudar en tratamientos de henfermedades raras ,como por ejemplo tmau
Muy interesantes estos nuevos avances científicos en genética molecular. Es posible disponer del artĺculo?
https://www.cell.com/cell/fulltext/S0092-8674(21)00353-6
De verdad acabo de leer la noticia y me he quedado sin palabras, padezco hiperfantasia y aun así no logro imaginar todas las posibilidades que ouede ofrecer esto y el campo de investigación que se abre:
-Tratamiento de rechazos de trasplantes
-Tratamiento de canceres
-Tratamiento de la senescencia
-Linfocitos de diseño
-Tratamiento de infecciones agudas
Esto es un antes y un después!
Creo que esto puede mejorar la activación y desactivación de los factores de Sinya Yamanaka e incluso también para las microesferas ANE de Chad A. Mirkin introduciendo trozos palindromicos del ARN del virus covid 19…
Es lo que están haciendo.
Genial está ciencia llevada de la mano de la ética y el bien común
Debería existir en España una política y un marcó legislativo que potenciara la ciencia y no el éxodo de cerebros como pasa hoy en día. Todo este tipo de investigación es nuestro futuro y además, al menos para mí, es fascinante (y todo lo relacionado con CRISPR más!!! ) . Gracias por divulgar ciencia.
Ya lo decía, si es posible «desmetilar» con «nutrientes celulares naturales» aunque sea en forma lenta, imaginemos lo que va a poder lograrse con esta TECNOLOGÍA.
Gracias a los científicos dedicados a darnos estás aportaciones.
Cada vez me maravilla más el mundo CRISPR.
Estoy leyendo su libro «Editando genes recorta, pega y colorea», aprendiendo y disfrutando con cada una de sus páginas.
Desde mi humilde posición de profesor del ciclo superior de FP «Laboratorio Clínico y Biomédico» en un instituto de Burgos, procuro trasladar a mis alumnos los conocimientos que con tanta claridad y pasión divulga a través de este blog.
Muchísimas gracias
Muchas gracias Javier! Me alegro que este blog te proporcione material para tus alumnos. Esa es mi intención! Un abrazo
crisper es el futuro