En junio del año pasado publiqué un artículo en el blog de la Asociación de Comunicadores de Biotecnología comentando los sorprendentes e increíbles resultados que unos investigadores estadounidenses habían reportado la semana anterior en la revista científica Nature Methods. Titulé a mi artículo «Biotecnología y post-verdad«, pretendiendo con ello resaltar eufemísticamente una noticia que tenía toda la pinta de no ser verdad, vamos, de ser mentira. Los investigadores mencionados habían generado un modelo animal, un ratón, mediante edición genética con las herramientas CRISPR, para sus estudios sobre retinosis pigmentaria, una grave enfermedad degenerativa de la retina de origen genético que acaba determinando la pérdida de visión de las personas afectadas. La generación del ratón ya la habían descrito en una publicación anterior, pero ahora se preguntaban, en este somero y reducido análisis, si tras la exposición del embrión del ratón a las herramientas de edición genética CRISPR eran capaces de detectar mutaciones en otros genes, distintos al planeado, pretendiendo con ello analizar el impacto real de las llamadas mutaciones no deseadas en secuencias genéticas similares (en inglés, off-target mutations).
El tema es ciertamente relevante, dado que la posibilidad, real, de alteración de genes similares es una de las limitaciones de los sistemas CRISPR de edición genética, quizás no la más importante, en mi opinión, y en la de otros muchos colegas. Sin embargo lo relevante es saber el impacto, el impacto real de estas posibles alteraciones, cómo de frecuentes pueden ser, antes de pensar en trasladar los beneficios derivados de la edición genética con CRISPR en modelos animales a las personas, antes de saltar a la clínica.
Los investigadores concluían su análisis relatando que habían encontrado centenares, miles de mutaciones inesperadas (unexpected mutations). Evidentemente, quizás sin proponérselo, la liaron parda con esas imprudentes conclusiones. De repente había un estudio, publicado en una revista de altísimo impacto, que rompía la baraja, que nos venía a decir que esto de pensar en utilizar las herramientas CRISPR en terapias génicas deberíamos ir quitándonoslo de la cabeza, tras sus resultados que aparentemente demostraban que la mera exposición de un embrión de ratón en desarrollo a la acción de las herramientas CRISPR (a la acción de la proteína Cas9, la endonucleasa dirigida por guías de ARN que inicia la cascada de acontecimientos en el genoma cortándolo en secuencias precisas) podía inducir la acumulación de múltiples mutaciones por doquier, en todo el genoma. Una verdadera pesadilla genética. El fin de las expectativas terapéuticas de las CRISPR. Rápidamente el artículo fue comentado en diversos medios generalistas. Y, naturalmente, las bolsas no tardaron en reaccionar, con caídas espectaculares inmediatas en el valor de las empresas biotecnológicas que están intentando comercializar las aplicaciones terapéuticas derivadas del uso de las herramientas CRISPR.
Pero algo olía mal desde el principio. Algo chirriaba en el estudio publicado. Los experimentos no parecían haberse realizado de la forma esperada, con el uso de adecuados controles, al comparar los genomas de los ratones editados genéticamente con los de otros ratones, no editados, usados como referencia. Ahí estaba la clave. La comunidad científica reaccionó de inmediato. No eran esos los resultados que teníamos muchos de nosotros, como los publicados en la misma revista por el grupo de Bill Skarnes, y también en mi laboratorio, en los que apenas encontrábamos, o directamente no se hallaban evidencias significativas de la presencia de mutaciones inesperadas. El tipo de reactivos CRISPR que los investigadores habían utilizado, con la persistencia de la proteína Cas9 mucho más allá de lo recomendable, tampoco parecía haber jugado a su favor, favoreciendo la oportunidad de cortar en secuencias distintas. Pero eso no podía explicar la magnitud de las mutaciones detectadas.
La primera evidencia de cuál podía haber sido el error la apuntó el genetista francés Gaetan Burgio, desde su laboratorio en Canberra (Australia). Resaltó el hecho sorprendente que los autores del estudio solamente hubieran secuenciado el genoma de DOS ratones editados con CRISPR y UN solo ratón control, aparente de la misma cepa genética, pero no relacionado con los anteriores. En efecto, por sorprendente que pueda parecer, por increible que pueda resultar que dicho estudio superara todos los supuestos filtros de revisión entre pares, y de selección por el comité editorial de la revista, todo este lío monumental (incluída la colosal pérdida de millones de euros por las empresas afectadas) se había montado a partir de los datos genómicos de solamente TRES ratones analizados. Adicionalmente, el ratón control no estaba relacionado con los ratones editados genéticamente, y podía ser genéticamente bastante distinto, como efectivamente así lo era, dando pie a suscitar la detección de múltiples mutaciones inesperadas al comparar su genoma con las secuencias de los genomas editados.
¿Cuál era pues la explicación más razonable? La explicación más plausible del «estudio» radicaba en un uso inadecuado de controles. Los investigadores habían usado como ratón control, como genoma de referencia, un ratón que creyeron era genéticamente idéntico a los fundadores editados pero que sin embargo no estaba relacionado, habían probablemente muchas generaciones de diferencia entre el ratón control y los editados. Y, debido a la deriva genética, ese ratón control podía haber acumulado cientos o miles de mutaciones espontáneas, distintas, pero nada relacionadas con la acción de las herramientas CRISPR, que luego aparecerían como mutaciones inesperadas, al realizar la comparación con el genoma de los ratones editados, usando programas bioinformáticos.
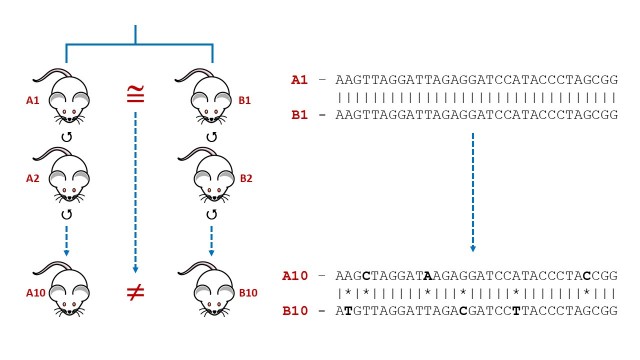
Sabemos de la existencia permanente, constante, de la deriva genética. En cada generación se acumulan entre 10 y 100 mutaciones espontáneas, debidas, entre otras causas, a la meiosis, durante la producción de los gametos (óvulos y espermatozoides) que darán lugar posteriormente al embrión. Asi pués, por muy estables genéticamente que creamos tener nuestros ratones, estos seguirán acumulando mutaciones en generaciones posteriores. En el esquema adjunto explico de forma gráfica el fenómeno de la acumulación de mutaciones espontáneas y de la deriva genética (en ingles genetic drift). Dos ratones, hermanos de camada, inicialmente genéticamente similares, prácticamente idénticos en su genoma, si los cruzamos con individuos de su misma cepa, pero de forma independiente, tras apenas unas 10 generaciones habrán acumulado y segregado mutaciones en distintos lugares del genoma. Si ahora volvemos a comparar la misma secuencia observaremos que ya no son idénticas. De la misma forma, si tuviéramos que comparar las secuencias de los ratones A1 y B1, en el ejemplo (que serían equivalentes con los ratones editados con CRISPR del estudio referido) con otro ratón «control», por ejemplo el ratón B10, observaríamos que tanto A1 como B1 tienen secuencias genéticas distintas a B10, que es exactamente lo que detectaron esos investigadores en su artículo de mayo de 2017.
Tras las dudas que resaltó Burgio diversos estudios insistieron en el uso de controles inadecuados como la explicación más sencilla para interpretar los resultados de los investigadores norteamericanos. Se publicaron respuestas muy críticas al estudio, también de científicos líderes en el campo de las CRISPR, como George Church, y otros vinculados a las empresas biotecnológicas implicadas. Entre estos estudios que rebatían las conclusiones del trabajo publicado en Nature Methods en 2017 destaca el concienzudo, preciso, pormenorizado y sistemático análisis, con los controles adecuados, realizado por otros investigadores del instituto Sanger, en Hinxton-Cambridge (Reino Unido), liderados por David Adams, que, aprincipios de febrero de 2018, demostraban, sin lugar a dudas y sin dejar espacio a interpretaciones alternativas, que comparando ratones editados y ratones control genéticamente relacionados (ratones progenitores, como controles, con los ratones hijos, como ratones editados, tras un experimento con CRISPR, lo que se denominan TRÍOS = padre+madre+hijo) eran incapaces de detectar mutaciones que pudieran estar causadas por las CRISPR en otros lugares del genoma de forma estadísticamente significativa a las variaciones genéticas esperadas, acumuladas tras cada generación. Punto final.
Tan solo quedaba que los autores retiraran el artículo inicial en Nature Methods. O que la revista, ante tal acumulación de evidencias en contra del estudio, optara por retirarlo como decisión editorial, mínimanente honrosa, admitiendo el error editorial de haber aceptado publicar ese estudio, y de alguna manera disculpándose por el terremoto mediático y científico que provocaron (la revista publicó, eso sí, un editorial en el que compartían la preocupación suscitada por ese estudio). Cualquiera de las dos soluciones hubiera sido la más adecuada. Sin embargo, nada de esto ha ocurrido. Lo que ha sucedido, lo que me ha invitado a escribir este nuevo comentario, es la aparición de un nuevo trabajo de los mismos autores del estudio de 2017, que acabamos de conocer esta semana.
En este nuevo estudio de título sugerente («Corrigendum and follow-up…«, Corrección y continuación….) los autores abordan dos experimentos similares en ratones, de dos fondos genéticos distintos, y comparan el genoma de los ratones editados con CRISPR con ratones control más cercanos a los editados, sin ser estrictamente tríos, como el experimento realizado correctamente por los investigadores británicos. A pesar de (de nuevo!) no usar los controles estrictamente adecuados constatan que la mayor cercanía entre ratones editados y controles les impide detectar las miles de mutaciones inesperadas que habían reportado en su estudio del año pasado (¡claro!). Y no les queda más remedio que concluir la discusión de su nuevo manuscrito indicando que (traducción del inglés) «… estos resultados genómicos apoyan la idea de que, en casos específicos, las herramientas CRISPR-Cas9 pueden editar el genoma con precisión en un organismo sin conllevar la introducción de numerosas mutaciones no deseadas…» y que todo ello «… sugiere un camino para el uso clínico de la edición genética mediada por CRISPR…» (¡acabáramos!). Lo que en román paladino viene a ser envainársela. Donde dije digo, digo Diego. Y a otra cosa mariposa.
¿Quién se responsabiliza ahora del desaguisado? ¿Quién se disculpa ante las empresas biotecnológicas, ante la comunidad científica, ante las asociaciones de pacientes, ante todos a quienes este estudió asustó y pretendió poner fin a unas expectativas terapéuticas que todavía apenas estábamos descubriendo? De esto hablamos también cuando nos referimos a la investigación responsable. De la responsabilidad que adquirimos los investigadores al publicar determinados resultados. De la responsabilidad que adquieren las revistas al aceptar para su publicación y dar por buenos unos resultados que, a todas luces, nunca deberían haberse publicado.
Si alguien desea revisar o ampliar los conocimientos básicos de genética, de la aparición y acumulación de mutaciones espontáneas, de la deriva genética, recomiendo la consulta de este estupendo manual de genética de roedores que escribieron en español en 2003 mis buenos amigos y colegas (y excelentes genetistas) Fernando Benavides y Jean-Louis Guénet, y que se ofrece gratuitamente desde los archivos de la sociedad ISTT.
NOTA añadida el 30 de marzo de 2018: La revista Nature Methods acaba de retirar hoy la polémica publicación de Schaefer y colaboradores que publicaron el 30 de mayo de 2017 al considerar lo siguiente (traducido del inglés): «Esta publicación se retira porque las variantes genómicas observadas por los autores en dos ratones tratados con CRISPR no pueden atribuirse de forma concluyente a CRISPR-Cas9«. De los seis autores del estudio original dos autores están de acuerdo con la retirada, pero los cuatro restantes, incluida la primera autora y el autor principal responsable del estudio, no.
Este artículo lo publiqué inicialmente en el blog general de Naukas el 27 de marzo de 2018.

