
Lluís Montoliu, Centro Nacional de Biotecnología (CNB – CSIC)
Esta semana una nueva variante de la herramienta de edición genética CRISPR ha saltado a las primeras páginas de los periódicos, algo poco frecuente en noticias de ciencia. ¿Qué tiene de especial para haber despertado el interés de miles de investigadores?
Para responder a esta pregunta tengo que explicar un par de nociones básicas de genética molecular.
El ejemplo más acertado para ilustrar las capacidades de las herramientas CRISPR es una navaja suiza multiusos. Estas permiten desde pelar manzanas y atornillar hasta descorchar botellas.
En su versión más sencilla, una herramienta CRISPR está constituida por dos moléculas:
- Una proteína (Cas9), una nucleasa que corta el ADN en sus dos cadenas.
- Una pequeña molécula de ARN, el acido nucléico que actúa de intermediario entre el material genético que hay en el núcleo de la célula (ADN) y la producción de proteínas que ocurre fuera del núcleo, en el citoplasma de la célula.
Veamos qué hace cada componente.
El sentido de la vida
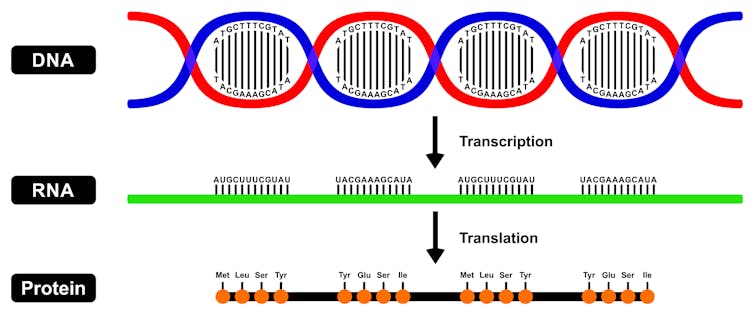
Lo normal es que la información genética progrese unidireccionalmente, desde el núcleo al citoplasma de la célula.
Una de las dos cadenas de ADN se copia en forma de ARN mediante un proceso que se llama transcripción. Este ARN sale al citoplasma y allí dirige la síntesis de una proteína determinada mediante un proceso que recibe el nombre de traducción.
Este flujo ADN -> ARN -> proteína es la base del funcionamiento de todas nuestras células.
Pero hace ya bastantes años se descubrió que existían unos virus, los retrovirus, que eran capaces de cambiar la dirección de ese flujo de información genética. Eran capaces de fabricar ADN a partir de ARN gracias a una nueva proteína que invertía el sentido de la ecuación. Dado que realizaba un proceso de transcripción al revés, se la bautizó como transcriptasa inversa.
Shutterstock/udaix
Cortar y pegar
En la versión más sencilla de CRISPR, la nucleasa Cas9 usa una pequeña molécula de ARN como guía para situarse en una posición concreta del genoma, sobre un gen determinado. Allí, tras realizar una última verificación, corta las dos cadenas de ADN.
Esto despierta los sistemas de reparación que se encargan de restaurar la continuidad del cromosoma. Por el camino obtenemos la edición o inactivación del gen deseado, según le aportemos o no un ADN molde que las proteínas reparadoras puedan usar para restaurar la secuencia.
Esto es la edición genética tradicional.
La tijera se convierte en lanzadera
¿Qué sucede si inhabilitamos la capacidad de corte de la Cas9 en una de las dos cadenas de ADN? Pues que solo cortará una de ellas. Esta Cas9 así modificada se llama nickasa y puede ser muy útil.
Si ahora inhabilitamos el corte de la otra cadena de ADN, la nickasa se convierte en una Cas9 muerta, incapaz de cortar el ADN. Pero seguirá localizándose en el lugar del genoma que la guía de ARN le indique: eso abre un mundo de oportunidades. Hemos convertido una tijera en una especie de lanzadera o módulo multiusos capaz de llevar la actividad que queramos a esa posición exacta del genoma. Bastará asociar esa nueva actividad a la nickasa o a la Cas9. El símil de la navaja multiusos cobra todo su esplendor.
El equipo de David Liu asoció, primero a una Cas9 inactiva y luego a una nickasa, una actividad denominada deaminasa, capaz de convertir una letra de la cadena de ADN en otra.
Con ello inventó en 2016 las variantes CRISPR llamadas “editores de bases”, capaces de cambiar determinadas bases del genoma de forma precisa. Con estos editores de bases se pensaba que podríamos tratar muchas enfermedades congénitas, al corregir las letras erróneas y substituirlas por las correctas, como si se tratara de un corrector molecular, como el famoso típex.
Sin embargo, su potencial quedó trastocado al descubrirse que se saltan el proceso de verificación de la secuencia sobre la cual se sitúan. Pueden ubicarse en muchos otros sitios del genoma, lo que produce numerosos cambios en genes que no deberíamos haber corregido y que darán resultados inesperados o no deseados.
Dos por el precio de uno
Esta semana Liu y sus colaboradores nos han vuelto a sorprender con su último trabajo publicado en la revista Nature. Esta vez han asociado una actividad transcriptasa reversa a una nickasa. En otras palabras, tenemos una proteína capaz de copiar ADN a partir de ARN en un sitio determinado del genoma.
¿Para qué podría servir? Pues para dirigir la copia de ADN que queremos producir según la información que contiene el ARN que actúa como molde.
¿Cómo hace para que el ARN actúe como molde? Muy sencillo. Se les ocurrió extender la pequeña molécula de ARN guía, que sirve para posicionar la nickasa en un sitio del genoma, y convertirla en una molécula bastante más larga. Ahora ese nuevo extremo puede usarse como molde para la otra cadena del ADN.
Eso es una propuesta muy inteligente que usa una misma molécula de ARN para dos cosas:
- Un extremo sirve para aparearse con una de las dos cadenas de ADN y así posicionar la Cas9 en el lugar deseado del genoma.
- El otro extremo sirve de molde para dirigir la síntesis de la otra cadena de ADN, la que hemos cortado. Podemos dirigir la síntesis a partir de la secuencia que le pongamos en ese nuevo extremo del ARN.
Así se pueden incorporar las letras correctas para corregir una mutación. O, al revés, generarla si se trata de saber qué pasa cuando ese gen está mutado.
Edición de calidad
A esta nueva capacidad de las herramientas CRISPR la han denominado “prime editing” (PE), que en inglés juega con el doble significado de “edición de calidad” y “guiada por un molde”.
Según sus autores, en teoría, se podrían corregir hasta un 89 % de los más de 75 000 errores genéticos que causan enfermedades en seres humanos. Estoy seguro de que ahora entienden mejor el grado de excitación que tenemos los investigadores con este nuevo “juguete”.
Lo que sabemos por ahora de las variantes PE es que funcionan en células humanas en cultivo, aunque no igual de bien con todos los tipos celulares. Se logran los cambios deseados con una buena eficiencia y, lo que es mejor, se reduce muchísimo la variabilidad de los resultados y la generación de mutaciones no deseadas en otras partes del genoma.
Pero todavía no sabemos si funcionará en animales y en personas. Tras la euforia inicial toca arremangarse. Muchos laboratorios de todo el mundo intentarán confirmar las buenas expectativas y con los nuevos experimentos iremos ampliando su potencial y descubriendo sus limitaciones.
Hay que celebrar esta nueva herramienta y felicitar a Liu y a sus colaboradores por su talento para combinar dos actividades (nickasa y transcriptasa inversa) que ni la evolución había asociado anteriormente. También ser prudentes: todavía no hemos curado ninguna enfermedad y puede que tardemos en hacerlo.![]()
Lluís Montoliu, investigador en Biología Molecular y Celular, Centro Nacional de Biotecnología (CNB – CSIC)
Este artículo fue publicado originalmente en The Conversation. Lea el original.
Este artículo lo publiqué inicialmente en The Conversation el 23 de octubre de 2019.

